화합물의 골격구조를 활용한 Transformer 기반 새로운 분자 설계
Copyright ⓒ 2025 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
전통적인 신약 개발은 새로운 약물을 시장에 출시하기까지 많은 시간과 막대한 비용이 소요되며, 높은 실패율로 인해 효율성이 낮다는 문제가 있다. 이러한 문제를 해결하기 위해 생성 모델을 활용한 혁신적인 접근법이 주목받고 있다. 본 연구에서는 트랜스포머 디코더 구조를 기반으로 화합물의 구조 정보를 문자열로 학습하여 새로운 화합물 구조를 생성하는 모델을 제안한다. 특히, 화합물에서 추출한 골격 구조(scaffold)를 임베딩하여 모델 입력에 포함함으로써, 결합 및 원자 정보와 골격 구조를 동시에 처리하였다. 벤치마크 데이터셋을 사용한 평가 결과, 골격 구조 임베딩을 적용한 모델이 데이터셋 별로 유효성 지표에서 0.964, 0.986의 우수한 성능을 보였다. 본 연구는 분자 생성 모델에 골격 구조 임베딩을 도입함으로써, 화학적 규칙을 준수하는 분자를 효과적으로 생성할 수 있는 방법을 제시하였으며, 신약 개발 분야에서 AI 기반 분자 설계의 효율성을 높이는 데 기여할 것으로 기대된다.
Abstract
Traditional drug development requires significant time and substantial costs in introducing a new drug to the market, and the high failure rates result in low efficiency. To address these challenges, innovative approaches utilizing generative models have garnered attention. In this study, we propose a model based on a transformer decoder architecture that learns structural information of compounds in string form to generate new compound structures. Specifically, by embedding scaffolds extracted from compounds into the model input, bond and atom information are simultaneously processed along with scaffold structures. Evaluation using benchmark datasets demonstrated that the model with scaffold embedding achieved superior performance in terms of validity metrics, recording 0.964 and 0.986 for each dataset. By introducing scaffold embedding into the molecule generation model, this study provides an effective way for generating molecules that adhere to chemical constraints, and is thus expected to contribute to improving the efficiency of artificial intelligence (AI)-based molecular design in the field of drug discovery.
Keywords:
Artificial Intelligence, Generative Model, Compound, Novel Molecular Design, Drug Discovery키워드:
인공지능, 생성형 모델, 화합물, 새로운 분자 설계, 신약 개발Ⅰ. 서 론
전통적인 신약 개발 과정은 연구개념 단계부터 임상 시험, 승인까지 평균 10~15년의 긴 시간과 수십억 달러의 비용이 소요되는 복잡한 과정이다[1]. 초기 단계에서의 합성 화합물 설계와 생물학적 활성 평가는 높은 비용과 시간을 요구한다. 또한, 수천가지의 후보 물질이 합성되더라도, 그 중 극히 일부만이 생물학적 활성이나 안전성을 확보하여 임상 시험 단계로 진입하게 된다[2]. 임상 시험 단계에서도 약물의 효능이나 안전성에 대한 문제가 발견되어 탈락하는 경우가 많아, 전체 개발 과정에서 실패하는 약물의 비율이 매우 높다. 이러한 높은 실패율은 신약 개발의 불확실성을 증가시키며, 개발 비용을 상승시키는 요인으로 작용한다. 전통적인 신약 개발 방식의 한계를 극복하기 위해서는 효율적이고 혁신적인 접근 방식과 기술의 도입이 필요하다.
인공지능 (AI; artificial intelligence) 은 기존의 신약개발 방식의 한계를 극복할 수 있는 수단 중 하나로 부각되고 있다[3]. 신약개발과 구성 분자 연구는 기본적으로 원하는 특성 profile을 가진 분자 구조를 설계하는 원리를 바탕으로 한다[4]. 그러나 새로운 분자에 대한 화학적 탐색 공간은 매우 방대하여 어려운 과제로 존재한다. 잠재적인 약물 유사 분자의 수는 1023~1060 개로 추정되며, 지금까지 합성된 분자는 약 108 개에 불과하다[5]. AI는 방대한 화학공간의 탐색을 가속화하여 후보물질을 도출하는 데 드는 시간과 비용을 줄일 수 있다. 대량의 화학 데이터와 생물학적 데이터를 분석하여 유망한 후보물질을 빠르게 식별하고, 그 특성을 예측함으로써 시행착오를 줄일 수 있다. 이러한 AI의 장점을 활용하여, 분자 설계에 생성 모델을 활용하려는 연구가 활발하게 진행중이다[3]. 데이터의 패턴을 학습하여 새로운 데이터를 생성하는 능력을 바탕으로, 컴퓨터비전과 자연어처리와 같은 데이터 분포를 모델링하는데 큰 발전을 일으켰다.
본 연구에서는 화합물의 구조적 정보를 텍스트 형태로 학습하여, 이를 기반으로 완전히 새로운 구조의 화합물을 생성하는 생성모델을 개발하였다. 제안된 모델은 트랜스포머(transformer) 기반의 디코더(decoder) 아키텍처를 채택하였으며, 입력 데이터를 모델에 적합한 형태로 전처리하는 과정을 거쳤다[6]. 구체적으로, 화합물로부터 골격 구조(scaffold) 정보를 추출하여 임베딩(embedding) 함으로써, 화합물의 세부 정보와 골격 구조 정보를 병렬적으로 처리하였다. 학습과정에서는 다음 토큰의 확률 분포와 실제 토큰 간의 손실(loss)을 최소화 하는 방식으로 진행되었으며, 이는 시퀀스-투-시퀀스(sequence-to-sequence) 학습 방법론을 따른다. 예측된 토큰은 이전에 생성된 토큰들에 기반한 예측 확률 분포의 결과이며, 이 과정을 반복함으로써 새로운 분자 구조를 생성한다. 본 논문은 2장에서 연구에 활용된 데이터셋과 전처리과정, 모델의 전반적인 구조와 학습과정에 대해 서술한다. 3장에서는 생성된 분자를 성능평가 지표를 통해 결과를 비교하고 4장에서는 결론을 서술하는 방식으로 진행한다.
Ⅱ. 화합물의 골격구조 기반 새로운 분자 설계
본 연구에서는 GuacaMol, MOSES (Molecular Sets) 총 두 가지 데이터셋을 활용하였으며 생성형 AI 모델을 통해 새로운 분자를 생성하고 이에 대해 성능을 평가하는 연구를 진행하였다[7],[8]. 분자 표기법은 SMILES (Simplified molecular-input line-entry system) 형태를 사용하였으며, 생성모델은 트랜스포머 모델의 디코더 구조를 활용하였다[9].
2-1 데이터셋
생성모델 학습을 위한 데이터로 벤치마크 데이터셋인 GuacaMol dataset을 활용하였다. GuacaMol은 ChEMBL 데이터베이스에서 추출된 1,591,378개의 분자를 포함하는 데이터셋이다[10]. 추출 과정은 크게 4개의 필터링 프로세스로 구성된다. salts를 제거하고, charge를 중화시키고, 100자 이상의 SMILES 문자열이 존재하는 분자는 제거하였다. 추가로 H, B, C, N, O, F, Si, P, S, Cl, Se, Br, and I 이외의 원소를 포함하는 분자를 제거하였다.
MOSES는 Zinc 데이터베이스에서 추출된 약 190만개의 소분자 화합물로 구성되어있다[11]. 250~350 달톤 사이의 분자량, 회전가능한 결합수가 7 미만, 3.5보다 작은 XlogP로 필터링 되었다. 또한 C, N, S, O, F, Cl, Br, H 이외의 원자를 포함하는 분자를 제거하고, 양이온이나 음이온과 같은 전하를 가진 원자를 포함하는 분자를 제거하였다. 마지막으로 cycle이 8개 이상의 원자로 구성된 분자를 제거하였다.
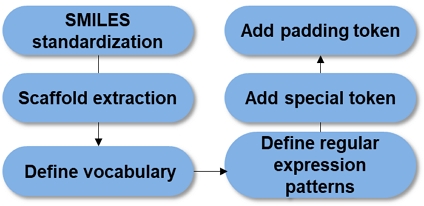
본 연구에서 벤치마크 데이터셋을 총 6단계의 전처리과정을 다음과 같이 수행하였다. 첫번째로, SMILES 문자열 표준화 진행 및 중복을 제거하였다. SMILES 문자열은 동일한 분자 구조를 다양한 방식으로 표현할 수 있기 때문에, 생성모델의 학습 효율성을 높이기 위해 SMILES 표준화 및 중복 제거가 필수적이다. RDKit을 활용하여 각 SMILES 문자열을 분자 객체로 변환하고, 표준화된 SMILES 문자열을 생성하였다[12]. 이 과정에서 분자의 구조를 표준화된 형태로 재구성하여 동일한 분자가 항상 동일한 SMILES 문자열로 매핑되도록 한다. 두번째로 각 SMILES 별로 scaffold를 추출하였다. scaffold란 화합물의 기본 골격을 의미하며, 이는 분자의 핵심 구조를 나타낸다. Bemis-Murcko scaffold는 가장 일반적으로 사용되는 scaffold 중 하나로, 분자의 ring과 결합을 포함한 핵심 골격을 추출한다. 분자가 단순한 선형 구조를 가지거나, 기본 골격을 형성할 수 없는 경우에는 scaffold를 추출하지 못하는데, 이러한 데이터는 제거하였다. 세번째로 어휘사전을 정의하였다. 어휘사전이란, 생성모델이 SMILES 문자열을 처리하고 생성하기 위해 사용하는 고유한 문자 집합을 의미한다. 본 연구에서는 SMILES 문자열을 구성하는 모든 고유 문자를 수집하고, 이를 순차적으로 인덱싱하여 고유한 토큰 집합을 형성하였다. 네번째로 정규표현식 패턴을 정의하고 토큰화를 진행하였다. 이는 SMILES 문자열을 의미있는 단위(token)으로 분리하는 과정이다. SMILES는 다양한 화학기호와 결합방식을 포함하고 있어, 이를 올바르게 토큰화하는 것이 모델의 성능에 직접적인 영향을 미친다. 자연어 처리과정에서 단어별로 토큰화를 진행하듯이, 사전에 정규 표현식 패턴을 정의하고 이를 기반으로 SMILES 문자열을 토큰화한다. 다섯번째로 special token을 추가하였다. 분자의 시작과 끝을 명확히 하기 위해 [SOS] (start-of-sequence)와 [EOS] (end-of-sequence) 토큰을 추가하여 모델이 분자 학습 및 생성의 시작과 끝을 인식할 수 있도록 설계하였다. 마지막으로 시퀀스의 길이를 일관되게 유지하도록 패딩토큰을 추가하였다. 학습에 사용되는 데이터를 정규표현패턴식을 통해 토큰화했을 때 가장 시퀀스 길이가 긴 데이터에 맞추어 패딩을 진행하였다. 패딩 토큰은 특별한 의미를 가지지 않으며, 시퀀스의 남은 부분을 채우기 위한 용도로 사용된다. 본 연구에서 사용된 데이터셋에 대한 통계는 표 1에 명시되어 있다. 데이터 전처리 과정은 그림 1에 나타나 있다.
2-2 모델 구조 및 워크플로우
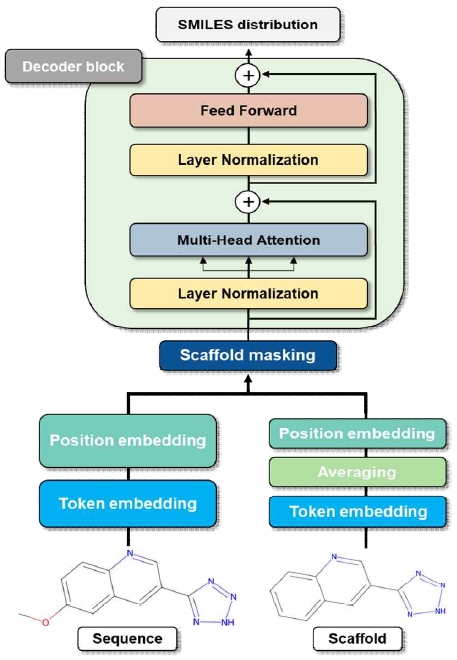
생성모델은 기본적인 트랜스포머 디코더 구조를 바탕으로 하며, 분자 생성에 특화된 모듈을 추가하여 SMILES 시퀀스를 효과적으로 생성하도록 설계하였다. 그림 2는 본 모델에서 사용된 생성모델의 전체 구조를 나타낸다. 모델은 임베딩 레이어와 다수의 디코더 블록으로 구성된다. 임베딩 레이어에서는 입력된 SMILES 시퀀스와 scaffold 정보를 모델 입력에 적합한 형태로 변환한다. 토큰 임베딩을 통해 각 토큰 인덱스를 고차원 벡터로 변환하고, 포지션 임베딩을 통해 순서정보를 추가한다. 이는 입력시퀀스의 문맥과 구조를 효과적으로 표현할 수 있도록 한다. scaffold 임베딩은 입력 시퀀스에 추가적인 구조적 정보를 제공하기 위해 사용된다. scaffold 정보를 벡터화하고, 평균을 낸후 이를 입력시퀀스의 임베딩과 결합하는 방식을 사용했다. 또한, 모델의 어텐션 메커니즘에서 scaffold 마스크를 활용하여 scaffold와 시퀀스 간의 상호작용을 제어하였다. scaffold 마스크는 어텐션 스코어 계산 시 scaffold 영역과 시퀀스 영역의 토큰 간 상호작용에 가중치를 부여하거나 차단하는 역할을 한다. 구체적으로, 마스크는 배치 크기와 시퀀스 길이에 맞춰 생성되며, scaffold 내부 및 시퀀스 내부에서는 어텐션이 자유롭게 이루어지도록 허용하지만, scaffold와 시퀀스 간의 불필요한 어텐션은 제한한다. 이를 통해 모델은 scaffold 정보에 대한 중요도를 조절할 수 있으며, 필요한 경우 scaffold에 더 집중하거나 시퀀스 자체에 집중할 수 있도록 유도할 수 있다. 디코더 블록은 멀티헤드 어텐션과 피드 포워드 신경망, 레이어 정규화 및 잔차 연결로 구성된다. 각 디코더 블록에서 멀티헤드 어텐션은 입력 시퀀스의 각 위치가 다른 위치와 맺는 관계를 다양한 표현 공간에서 병렬적으로 학습하도록 한다. 어텐션과 각 헤드별 수식, 멀티헤드 어텐션의 수식은 각각 (1)-(3)에 표현되어 있다.
| (1) |
| (2) |
| (3) |
상단의 수식에서 공통적으로 Q는 쿼리, K는 키, V는 밸류를 나타낸다. 수식 (1)에서 dk는 키의 차원을 나타내며 softmax는 소프트맥스 활성화 함수를 의미한다. KT는 키의 전치행렬을 나타낸다. 수식 (2)에서 , , 는 각 헤드에 대한 학습 가능한 가중치 행렬을 나타내며 headi 는 i번째 어텐션 헤드를 의미한다. 수식 (3)에서 WO는 최종 출력으로 변환하기 위한 학습 가능한 가중치 행렬을 나타내며 h는 어텐션 헤드의 개수를 의미한다.
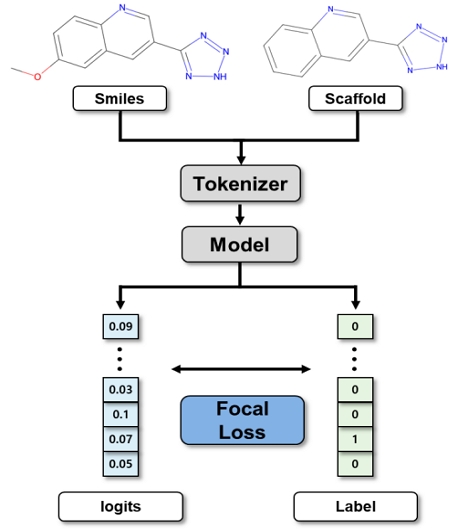
생성모델의 학습과정은 그림 3에 시각화되어 있으며, 입력 시퀀스 데이터와 라벨 데이터의 처리를 중심으로 이루어진다. 입력 시퀀스 데이터는 고정된 길이의 시퀀스로 패딩처리가 완료된 상태이며, 이는 모델이 일관된 입력 길이를 유지하도록 한다. 라벨 데이터는 입력 시퀀스 데이터에서 첫 번째 토큰을 제거하고, 시퀀스의 끝에 패딩 토큰을 추가하여 생성된다. 이 과정은 모델이 다음 토큰을 예측하도록 학습시키기 위한 것으로, 시퀀스 내의 각 위치에서 다음에 올 토큰을 정답으로 설정한다. 즉 입력 시퀀스 에 대해 라벨 데이터는 로 구성된다. 모델은 입력 시퀀스를 처리하여 SMILES 토큰에 대한 로짓(logits) 값을 출력한다. 이 로짓은 모델의 최종 출력 레이어에서 계산되며, 각 위치에서 다음에 올 토큰에 대한 예측 점수를 나타낸다. 로짓의 차원은 어휘사전의 크기와 동일하여, 모델이 학습된 모든 토큰에 대한 예측을 수행할 수 있다.
학습과정에서 모델은 출력된 로짓 값과 실제 라벨 데이터를 비교하여 손실값을 계산한다. 손실함수로는 focal loss를 사용하여 클래스 불균형 문제를 완화하고 어려운 샘플에 대한 학습을 강화하였다. focal loss는 기존의 교차 엔트로피 손실에 조정된 가중치를 부여하여 잘 분류되는 쉬운 샘플의 영향력을 감소시키고, 어려운 샘플에 더 큰 가중치를 부여한다. 분자 생성과 학습에서는 특정 토큰이나 패턴이 빈번하게 등장할 수 있는데, focal loss는 이러한 불균형을 완화시켜 균형잡힌 학습을 할 수 있게 한다. 수식은 (4)에 표현되어 있다.
| (4) |
pt 는 예측 확률 값이며 예측값과 실제 클래스간의 확률을 나타낸다. α는 가중치 값으로, 불균형한 소수 클래스에 더 큰 가중치를 줄 때 사용된다. γ는 초매개변수로, 잘 분류된 샘플에 대해 가중치를 줄이고, 잘못된 샘플에 대해 가중치를 높이는 역할을 한다.
데이터셋별로 어휘사전 크기나 최대 시퀀스 길이가 다르기 때문에 각각 실험을 진행하였으며, 적용한 하이퍼파라미터는 표 2와 같다.
생성모델로 SMILES 시퀀스를 생성하기 위해서는 초기토큰과 scaffold 조건을 사전에 정의해야한다. 생성과정은 시작토큰인 [SOS] 토큰과 반영하고자 하는 scaffold 조건을 모델의 입력으로 제공하는 것으로 시작한다. 모델은 입력된 시퀀스를 기반으로 다음에 올 토큰의 확률 분포를 예측하며, 이는 모델의 출력에 소프트맥스 함수를 적용하여 각 토큰에 대한 확률을 얻는다. 이 확률 분포에 따라 다음 토큰을 샘플링하며, 선택된 토큰은 현재 시퀀스에 추가된다. 확장된 시퀀스는 다시 모델의 입력으로 사용되어 다음 토큰 예측에 활용된다. 이러한 과정은 시퀀스 종료 토큰인 [EOS]가 생성되거나, 생성된 시퀀스의 길이가 사전의 정의된 최대 길이인 100에 도달할 때 까지 반복된다. 생성이 완료되면, 모델이 출력한 토큰 인덱스 시퀀스는 미리 정의된 어휘 사전을 통해 인덱스에서 토큰으로 변환되고, 이를 다시 결합하여 최종적인 SMILES 문자열을 얻는다.
Ⅲ. 실험 및 결과
3-1 모델 성능평가 지표
본 연구에서 제안한 모델의 성능 평가는 기존의 회귀모델 및 분류 모델과는 차별화된 접근 방식을 채택하여, de novo 분자 설계 분야에서 널리 사용되는 표준 성능 지표를 기반으로 수행되었다. 평가에 사용된 주요 성능 지표는 유효성(validity), 고유성(uniqueness), 신규성(novelty) 으로 구성되었다. 각 지표는 모델의 생성 능력을 다각도로 평가하는 데 중요한 역할을 한다.
유효성의 경우, 생성된 분자가 실제로 화학적으로 유효한지를 평가하는 지표로, RDKit 라이브러리를 기반으로 수행되었다. 본 연구에서는 총 10,000개의 분자를 생성하고, 이 중 화학적으로 유효한 구조를 가진 SMILES 문자열의 비율로 측정하였다. 수식 (5)에서는 생성된 분자들 중 유효한 화학 구조를 가진 문자열의 집합을 V 로 표기하였다. 고유성은 생성된 유효한 분자들 중 고유한 분자의 비율을 나타내며, 이는 모델이 다양한 분자를 생성할 수 있는지를 평가하는 지표이다. 고유성은 수식 (6)과 같이 계산되며, 고유한 분자 집합을 U 로 표기하였다. 고유성이 낮을 경우, 이는 모델이 반복적인 분자 생성을 수행하고 있음을 의미하며, 모델의 분포 학습 수준이 낮음을 시사한다. 신규성은 학습 데이터셋에 존재하지 않는 유효한 고유 생성 분자의 비율을 나타낸다. 이는 모델이 학습 데이터에 과적합되지 않고, 새로운 화합물을 창출하는 능력을 평가하는 지표이다. 수식 (7)에서는 학습 데이터셋에 없는 유효한 고유 생성 분자 집합을 T 로 표기하였다. 신규성이 낮다는 것은 모델이 학습 데이터셋에 과적합 되었음을 의미하며, 이는 모델이 새로운 분자를 표과적으로 생성하지 못하고 기존 데이터에만 의존하고 있음을 나타낸다.
| (5) |
| (6) |
| (7) |
3-2 모델별 성능 비교
GuacaMol 데이터셋과 MOSES 데이터셋을 학습한 각각의 모델에 대해 성능평가를 실시하였으며 결과는 표 3과 같다. 모델은 scaffold 임베딩을 적용한 경우와 적용하지 않은 경우로 나누어 테스트 데이터를 적용하여 비교 분석하였다.
GuacaMol 데이터셋에서 scaffold 임베딩을 적용한 모델은 유효성 0.964, 고유성 0.957, 신규성 1.0의 성능을 보였다. 반면, scaffold 임베딩을 적용하지 않은 모델은 유효성 0.663, 고유성 0.975, 신규성 1.0을 기록하였다. scaffold 임베딩을 적용한 모델은 적용하지 않은 모델에 비해 유효성이 0.3 이상 높게 나타났다. 고유성 측면에서는 scaffold 임베딩을 적용하지 않은 모델이 약 0.018 높은 수치를 보였으나, 두 모델 모두 0.95 이상의 높은 수준의 고유성을 유지하였다. 신규성은 두 모델 모두 1.0로, 새로운 구조의 분자를 생성하는 데에 있어 차이가 존재하지 않았다.
MOSES 데이터셋에서 scaffold 임베딩을 적용한 모델은 유효성 0.986, 고유성 0.927, 신규성 0.922의 성능을 나타냈다. scaffold 임베딩을 적용하지 않은 모델은 유효성 0.819, 0.912, 신규성 0.983을 기록하였다. 이 결과에서도 scaffold 임베딩을 적용한 모델이 유효성에서 0.167 높은 성능을 보였다. 고유성 측면에서는 scaffold 임베딩을 적용한 모델이 0.015 높은 수치를 기록하였다. 그러나 신규성에서는 scaffold 임베딩을 적용하지 않은 모델이 0.061 더 높은 수치를 기록하였다.
scaffold 임베딩을 적용한 모델은 유효성과 고유성 측면에서 우수한 성능을 보였다. 유효성의 향상은 모델이 scaffold 정보를 활용하여 화학적 규칙을 보다 잘 준수하는 분자를 생성할 수 있게 되었기 때문으로 해석된다. 또한, scaffold 정보를 활용하는 것이 화학적으로 타당한 분자를 생성하는 능력을 향상시켰음을 시사한다. 고유성의 향상은 scaffold 임베딩이 다양한 구조의 분자를 생성하는 데 기여했음을 나타낸다. 반면, 신규성 측면에서 scaffold 임베딩을 적용하지 않은 모델이 더 높은 수치를 보이는 경우도 있는데, 이는 scaffold 없이 학습된 모델이 더 다양한 새로운 구조를 탐색할 가능성을 보유하기 때문이다. 그러나 실용적인 분자 설계에서는 화학적 유효성과 고유성이 더 중요하게 고려되는 경우가 많다. 또한, 유효한 분자의 집합이 적다면, 고유성과 신규성을 추론하는 과정에서 분자의 모수가 작아지므로, 값이 크게 도출되는 가능성이 존재할 수 있다.
3-3 생성된 분자의 특성 평가
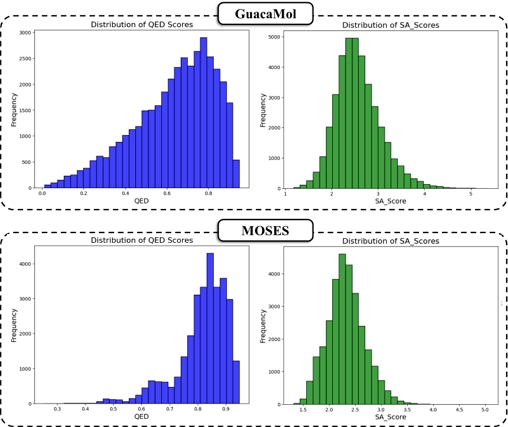
생성된 분자의 실제 화학적 특성을 평가하기 위해 QED (quantitative estimate of drug-likeness) 점수와 합성가능성 점수를 계산하였다. QED 점수는 분자가 약물 후보가 될 가능성을 0에서 1 사이의 값으로 평가한다[13]. 합성 가능성 점수는 분자의 합성 난이도를 1에서 10 사이의 값으로 평가한다[14]. QED 점수가 높을수록 생성된 분자의 약물 유사성이 높음을 의미하며, 합성 가능성 점수가 낮을수록 분자의 합성이 더 용이함을 나타낸다. 그림 4는 각 데이터셋으로 학습한 모델이 생성한 분자들의 QED 점수와 합성 가능성 점수의 분포를 보여준다.
Distribution of the quantitative estimate of drug-likeness (QED) and synthetic accessibility scores for generated molecules
GuacaMol 데이터셋으로 학습한 모델이 생성한 분자의 QED 점수는 최소 0.10에서 최대 0.94까지 분포하였다. 또한 전체 분자의 약 62%가 QED 점수 0.6 이상을 기록하였고, 약 21%가 0.8 이상을 기록하였다. 생성된 분자의 합성 가능성 점수는 최소 1.17에서 최대 5.37까지 분포하였다. 전체 분자의 약 82%가 합성 가능성 점수 3 이하를 기록하였으며, 99%가 5 이하의 점수를 보였다.
MOSES 데이터셋으로 학습한 모델이 생성한 분자의 QED 점수는 최소 0.25에서 최대 0.94까지 분포하였다. 약 97%의 분자가 QED 점수 0.6 이상을 기록하였고, 약 67%가 0.8 이상을 기록하였다. 합성 가능성 점수는 최소 1.31에서 최대 5.06까지 분포하였다. 약 96%의 분자가 합성 가능성 점수 3 이하를 기록하였으며, 모든 분자가 5 이하의 점수를 보였다.
Ⅳ. 결 론
신약 개발 과정은 많은 시간과 비용이 소요되며, 수많은 후보 물질 중 극히 일부만이 최종적으로 약물로 승인되는 높은 실패율을 보인다. 이러한 문제를 해결하기 위해 인공지능(AI)을 활용한 효율적이고 혁신적인 접근 방식이 필요하다. 특히, AI 기반의 생성 모델은 방대한 화학적 탐색 공간을 효과적으로 탐색하여 새로운 후보 물질을 설계하는 데 기여할 수 있다.
본 연구에서는 트랜스포머 기반의 디코더 구조를 활용하여 새로운 화합물 구조를 생성하는 생성 모델을 제안하였다. 특히, 화합물로부터 추출한 scaffold 정보를 임베딩하여 모델 입력에 포함해서 활용함으로써, 화합물의 세부 정보와 골격 구조 정보를 병렬적으로 처리하는 방식을 진행하였다. 모델은 출력된 토큰의 확률 분포와 실제 토큰 간의 손실을 최소화하는 방식으로 학습하였다. 모델의 성능은 벤치마크 데이터셋을 활용하여 평가하였으며 유효성, 고유성, 신규성 지표를 사용하였다. 실험 결과, scaffold 임베딩을 적용한 모델은 scaffold를 활용하지 않은 모델에 비해 유효성 측면에서 우수한 성능을 보였다. 이러한 결과는 골격 구조 임베딩의 적용이 생성 모델의 성능 향상에 효과적임을 나타낸다. 특히, 유효성의 향상은 모델이 화학적 규칙을 준수하는 분자를 생성하는 능력이 향상되었음을 의미한다.
본 연구를 통해 분자 생성 모델에 골격 구조 임베딩을 도입함으로써 신약 개발의 효율성과 혁신성을 높일 수 있는 가능성을 제시하였다. 향후 연구에서는 본 모델의 구조를 개선하고, 추가적인 화학적 특성이나 생물학적 활성 정보를 반영하여 생성되는 분자의 품질과 실용성을 높이는 방향으로 나아갈 수 있다. 또한, 생성된 분자의 약리학적 특성(pharmacological properties) 을 고려한 평가를 통해 모델의 실용적인 적용 가능성을 검증하는 연구가 필요하다. 더 나아가, 다양한 데이터셋과의 비교실험을 통해 모델의 범용성을 확인하고, 다른 생성 모델과의 통합이나 하이브리드 모델 개발을 통해 성능을 극대화할 수 있을 것이다.
Acknowledgments
본 연구는 정부(과학기술정보통신부)의 재원으로 정보통신기획평가원의 지원을 받아 수행된 지역지능화혁신인재양성사업(IITP-2024-RS-2022-00156287)과 과학기술정보통신부 및 정보통신기획평가원의 학.석사연계ICT핵심인재양성사업의 연구(RS-2022-00156385), 그리고 식품의약품안전처의 연구개발비(RS-2024-00332003)로 수행되었으며, 관계부처에 감사드립니다.
References
-
N. Berdigaliyev and M. Aljofan, “An Overview of Drug Discovery and Development,” Future Medicinal Chemistry, Vol. 12, No. 10, pp. 939-947, 2020.
[https://doi.org/10.4155/fmc-2019-0307]

-
M. Olivecrona, T. Blaschke, O. Engkvist, and H. Chen, “Molecular De-Novo Design through Deep Reinforcement Learning,” Journal of Cheminformatics, Vol. 9, 48, September 2017.
[https://doi.org/10.1186/s13321-017-0235-x]

-
K.-K. Mak and M. R. Pichika, “Artificial Intelligence in Drug Development: Present Status and Future Prospects,” Drug Discovery Today, Vol. 24, No. 3, pp. 773-780, March 2019.
[https://doi.org/10.1016/j.drudis.2018.11.014]

-
J. You, B. Liu, R. Ying, V. Pande, and J. Leskovec, “Graph Convolutional Policy Network for Goal-Directed Molecular Graph Generation,” in Proceedings of the 32nd International Conference on Neural Information Processing Systems (NIPS ’18), Montréal, Canada, pp. 6412-6422, December 2018.
[https://doi.org/10.48550/arXiv.1806.02473]

-
P. G. Polishchuk, T. I. Madzhidov, and A. Varnek, “Estimation of the Size of Drug-Like Chemical Space Based on GDB-17 Data,” Journal of Computer-Aided Molecular Design, Vol. 27, pp. 675-679, August 2013.
[https://doi.org/10.1007/s10822-013-9672-4]

-
A. Vaswani, N. Shazeer, N. Parmar, J. Uszkoreit, L. Jones, A. N. Gomez, ... and I. Polosukhin, “Attention is All You Need,” in Proceedings of the 31st Conference on Neural Information Processing Systems (NIPS ’17), Long Beach: CA, pp. 6000-6010, December 2017.
[https://doi.org/10.48550/arXiv.1706.03762]

-
N. Brown, M. Fiscato, M. H. S. Segler, and A. C. Vaucher, “GuacaMol: Benchmarking Models for De Novo Molecular Design,” Journal of Chemical Information and Modeling, Vol. 59, No. 3, pp. 1096-1108, March 2019.
[https://doi.org/10.1021/acs.jcim.8b00839]

-
D. Polykovskiy, A. Zhebrak, B. Sanchez-Lengeling, S. Golovanov, O. Tatanov, S. Belyaev, ... and A. Zhavoronkov, “Molecular Sets (MOSES): A Benchmarking Platform for Molecular Generation Models,” Frontiers in Pharmacology, Vol. 11, 565644, December 2020.
[https://doi.org/10.3389/fphar.2020.565644]

-
D. Weininger, “SMILES, a Chemical Language and Information System. 1. Introduction to Methodology and Encoding Rules,” Journal of Chemical Information and Computer Sciences, Vol. 28, No. 1, pp. 31-36, February 1988.
[https://doi.org/10.1021/ci00057a005]

-
D. Mendez, A. Gaulton, A. P. Bento, J. Chambers, M. De Veij, E. Félix, ... and A. R. Leach, “ChEMBL: Towards Direct Deposition of Bioassay Data,” Nucleic Acids Research, Vol. 47, No. D1, pp. D930-D940, January 2019.
[https://doi.org/10.1093/nar/gky1075]

-
T. Sterling and J. J. Irwin, “ZINC 15 - Ligand Discovery for Everyone,” Journal of Chemical Information and Modeling, Vol. 55, No. 11, pp. 2324-2337, October 2015.
[https://doi.org/10.1021/acs.jcim.5b00559]

- RDKit. Open-Source Cheminformatics Software [Internet]. Available: https://www.rdkit.org/, .
-
G. R. Bickerton, G. V. Paolini, J. Besnard, S. Muresan, and A. L. Hopkins, “Quantifying the Chemical Beauty of Drugs,” Nature Chemistry, Vol. 4, No. 2, pp. 90-98, February 2012.
[https://doi.org/10.1038/nchem.1243]

-
P. Ertl and A. Schuffenhauer, “Estimation of Synthetic Accessibility Score of Drug-Like Molecules Based on Molecular Complexity and Fragment Contributions,” Journal of Cheminformatics, Vol. 1, 8, June 2009.
[https://doi.org/10.1186/1758-2946-1-8]

저자소개

2023년:전남대학교 대학원 (공학석사)
2023년~현 재: 전남대학교 지능전자컴퓨터공학과 석사과정
※관심분야:생명정보학(bioinformatics), 인공지능(artificial intelligence) 등

2012년:한국항공대학교 정보통신공학과 (공학석사)
2018년:한국과학기술원 바이오및뇌공학과 (공학박사)
2018년~2019년: 국민건강보험공단 빅데이터실 부연구위원
2019년~현 재: 전남대학교 지능전자컴퓨터공학과 교수
※관심분야:생명정보학(bioinformatics), 인공지능(artificial intelligence), 빅데이터(big data) 등