머신러닝 기반 약물 이상사례(KARES DB)분석을 통한 인체 장기 장애 예측
Copyright ⓒ 2024 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
사람이 약을 복용하면 약에 대한 부작용으로 인해 인체에 장기 기능 장애가 발생한다. 이러한 인체 내 약물 부작용으로 인한 장기 기능 장애 현상은 정상 상태에 비해 불균형한 상태로 데이터를 생성한다. 불균형 데이터를 이용하여 분석할 때, 머신러닝 모델에서는 클래스 불균형이라는 문제가 발생한다. 인체의 장기 기능 장애를 분석하기 위한 기계 학습 방법을 최초로 제안한다. 8개의 머신러닝 모델을 사용하여 분석한다. 두 가지 신체 질환에 대해 데이터 샘플링을 수행한 횟수와 샘플링 적용 여부에 따른 머신러닝 모델의 성능을 분석한다. 데이터 샘플링을 수행하지 않은 경우에 비해 데이터 샘플링을 적용한 경우 머신러닝 모델의 성능이 향상되는 것을 확인했다. 제안한 방법이 장기 기능 장애 발생 예측이 개선됨을 확인했다.
Abstract
When taking medication, organ dysfunction can occur in the human body due to adverse reactions to the drug. This organ dysfunction phenomenon generates data in an unbalanced state compared to the normal state. When analyzing unbalanced data using a machine learning model, the class imbalance problem occurs. We propose a machine learning method for analyzing organ dysfunction in the human body for the first time. We considered eight machine learning models and analyzed their performance according to the amount of data sampling and whether sampling was applied for two body diseases. We confirmed that the performance of the machine learning model improved when data sampling was applied. We confirmed that the proposed method improves the prediction of organ dysfunction occurrence.
Keywords:
Human Organs, Machine Learning, Class Imbalance, KAERS DB, Adverse Drug Cases키워드:
인체 내 장기, 머신러닝, 클래스 불균형, 약물 이상사례Ⅰ. 서 론
약물 이상 사례는 환자의 건강에 심각한 영향을 미칠 수 있는 중요한 의료 문제 중 하나이다. 약물 이상사례는 예상치 못한 반응으로 인해 약물 치료의 목적을 방해하며, 심각한 경우 입원 연장, 추가적인 치료, 심지어 사망에도 이를 수 있다. 이러한 약물 이상 사례는 환자뿐만 아니라 의료 시스템 전체에도 막대한 비용을 초래할 수 있다. 따라서 약물 이상사례를 조기에 예측하고 관리하는 것은 환자 안전과 의료 비용 절감에 중요한 역할을 수행한다. 이러한 약물 이상 사례에 대해서 사전에 아는 것은 중요하며 약물 이상 사례 중에서도 인체 내 장기의 장애 현상을 분석하는 것은 더욱 중요하다[1].
사람의 장기 장애 현상에서 소화기와 신장에 대해서 분석하는 것은 특히 약물 흡수와 배설 과정에서 중요한 역할을 수행하는 장기이기 때문에 매우 중요한 중요성을 가지고 있다[2]. 또한 장애 현상을 분석할 때에는 발생 빈도가 적기 때문에 불균형한 상태로 발생되는 경우가 많다. 머신러닝 모델을 이용하여 분석을 진행할 때에는 빈도가 높은 데이터에 대해서만 학습을 잘하기 때문에 빈도가 불균형한 상태로 발생된 데이터에 대해서는 모델의 성능이 잘 나오지 않는다는 문제가 있다. 즉, 많은 데이터에 대해서만 학습이 잘 이뤄지게 되는 문제점이 존재한다[3]. 따라서 약물 이상 사례에서 드물게 발생하는, 즉 발생 빈도가 적은 인체 내 장기의 장애 현상을 머신러닝 성능의 개선을 통하여 높은 정밀도를 지니게끔 분석할 필요성이 있다고 판단했다. 이에 기존의 통계적 기법 및 머신러닝 모델들의 한계를 극복하기 위해서 다양한 샘플링 기법을 적용하여 불균형 데이터를 효과적으로 처리하고, 예측 성능을 향상시키는 방법을 분석한다. 이어지는 제안 방법 섹션에서는 우리가 사용한 데이터와 제안된 머신러닝 모델에 대해서 구체적으로 설명한다. 본 논문의 공헌도는 아래와 같다.
처음으로 인체 내 장기의 장애 현상을 분석하는 머신러닝 방법을 제안한다.
장기의 장애 현상이 불균형하게 발생함을 고려하여, 이를 개선하기 위한 머신러닝 분석 방법을 제안한다. 이를 위해서 본 연구에서는 자발적 약물 이상사례보고시스템인 한국의약품부작용보고원시자료 시스템 데이터베이스(Korea Adverse Event Reporting System Database, KAERS DB)를 사용한다.
8가지 머신러닝 방법을 이용해서 성능을 분석한다. 머신러닝 모델의 평가 지표로는 Accuracy, F1score, Precision, Recall, 그리고 ROC AUC (Receiver and Operator Curve Area Under the Curve)를 이용하여 분석한다.
Ⅱ. 머신러닝 기반 약물 이상 사례 예측 연구
약물 이상사례 예측을 위한 연구는 오랜 기간 동안 지속되어 왔다. 초기의 연구들은 주로 통계적 모델링과 규칙 기반 시스템을 이용한 예측 방법에 초점을 맞추었다. 이러한 접근법은 특정 약물 또는 질환에 대한 예측에 유용할 수 있었으나, 복잡한 환자 데이터의 다양한 상호작용을 충분히 반영하지 못했다는 한계가 있다.
전통적인 통계적 접근법은 로지스틱 회귀(logistic regression)와 같은 회귀 모델을 주로 사용하였다. 이러한 모델은 특정 변수들이 약물 이상사례 발생에 미치는 영향을 평가하는 데 유용하였으나, 복잡한 비선형 관계를 처리하는 데 한계가 있다. Bate et al.[4]은 통계적 기법을 사용하여 약물 부작용을 분석하였으나, 복잡한 다변량 관계를 설명하는 데 어려움을 겪었다. 이는 특히 약물 간 상호작용이나 환자의 다변량 임상 특성들을 포함하는 상황에서 더 두드러졌다. 이러한 한계는 약물 이상사례 예측 모델의 성능을 제약하며, 실질적인 임상 적용에 어려움을 겪게 한다.
최근에는 머신러닝 기법이 이러한 한계를 극복하기 위한 대안으로 등장했다. Schuemie et al.[5]은 Electronics Health Records(EHR) 데이터를 활용하여 머신러닝 모델을 통해 약물 이상 사례를 예측하는 연구를 수행하였으며, 이러한 모델들이 통계적 모델보다 우수한 예측 성능을 보인다는 것을 입증했다. 특히, 앙상블 기법(예: Random Forest 그리고 AdaBoost)은 복잡한 데이터 구조를 효과적으로 처리할 수 있으며, 다양한 변수 간의 상호작용을 반영하여 예측 성능을 높일 수 있는 것으로 보고했다.
Liu et al.[6]은 약물-단백질 상호작용 정보를 활용하여 약물-약물 상호작용으로 인한 이상 약물 반응(Adverse Drug Reaction, ADR)을 예측하는 모델을 개발했다. 이 연구에서는 약물-단백질 상호작용 정보를 기반으로 약 800개의 약물에 대한 상호작용 프로파일을 구축하여 약물 조합이 ADR을 유발할 가능성을 평가했다. 그 결과 약물-약물 상호작용으로 인한 ADR을 예측하는 모델의 평균 정확도는 89%였으며, 현재 임상적으로 보고되지 않은 잠재적인 ADR도 예측할 수 있는 것으로 나타났다. 이 연구는 약물 상호작용으로 인한 ADR을 예측하고 이를 최소화할 수 있는 잠재적 약물 조합을 제시함으로써 임상 약물 사용의 안전성을 높이는 데 기여할 수 있음을 보여주었다.
Scholl et al.[7]의 연구에서는 약물 부작용(ADR)을 예측하기 위한 새로운 통계 모델을 개발하여 기존 모델보다 향상된 예측 성능을 달성했다. 이 연구는 자발적 보고 시스템에서 수집한 ADR 데이터를 기반으로 통계적 신호 감지 성능을 개선하기 위한 예측 모델을 개발하였다. 주요 예측 인자로는 보고 건수, 불균형성(Reporting Odds Ratio at the 2.5% lower confidence limit, ROR025), 나란조 점수, 제조업체 보고율(Marketing Authorization Holder Reporting Rate, MAH), 의료 전문가 보고율(Healthcare Professional Reporting Rate, HCP)이 포함되었다. 이 모델은 새로운 신호를 감지하는 데 도움이 되도록 약물과 ADR 간의 연관성을 우선시하였으며, 이전 모델에 비해 상위 800개 신호에서 신호 감지율이 58.2% 증가하는 결과를 보였다. 또한 해당 모델은 이전 모델보다 더 높은 AUC score (Area Under the Curve, 0.740)로 예측 성능이 향상되는 것으로 나타났다. Scholl et al.[7]의 연구는 ADR 예측 모델에 다양한 변수를 통합하여 신호 탐지 효율을 높일 수 있음을 보여주며, 다른 국가나 데이터베이스에 적용할 수 있음을 시사한다. 이 연구는 기존의 통계적 접근법을 보완하고, 머신러닝 기법을 활용하여 약물-약물 상호작용으로 인한 ADR을 보다 정교하게 예측하는 데 기여할 수 있다는 점에서 유용한 참고 자료가 될 수 있다.
심층 신경망(Deep Neural Networks, DNN)과 같은 딥러닝 기법은 비선형적인 데이터 패턴을 학습하는 데 강점을 보였다. Miotto et al.[8]은 DNN을 사용하여 대규모 EHR 데이터를 분석하였으며, 이 모델이 기존의 통계적 기법보다 약물 이상사례를 더 잘 예측할 수 있음을 보여주었다. 그러나 이러한 모델들은 종종 많은 데이터와 계산 자원을 요구한다. Qureshi et al.[9]의 연구는 인공지능을 활용하여 약물 개발 및 약물 이상사례 예측에서 임상적 적용 가능성을 높이는 데 초점을 맞추고 있다. 이 연구는 다양한 머신러닝 기법을 통합하여 약물의 안전성을 평가하고, 약물 부작용 발생 가능성을 예측하는 데 기여하고 있으며, 특히 신약 개발 과정에서 약물 부작용 예측 모델의 활용 가능성을 제시하고 있다.
Ⅲ. 약물 이상 사례에서 클래스 불균형 문제를 해결하기 위한 제안 방법
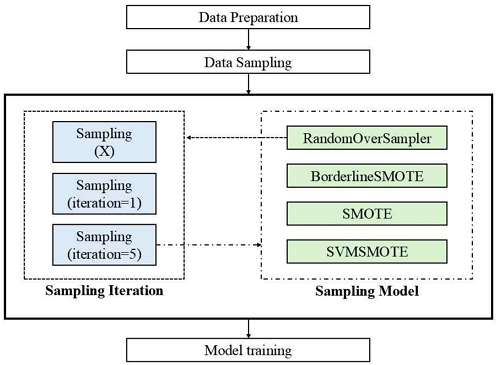
그림 1은 제안된 모델에서 사용하는 샘플링 기법에 대해 시각화 한 그림이다. 제안된 방법에서는 샘플링 횟수(수행하지 않은 건, 1회 수행한 건, 5회 수행한 건)와 사용된 모델의 종류(RandomOverSampler, BorderlineSMOTE, SMOTE, 그리고 SVMSMOTE) 에 따라 결과를 비교하게 된다.
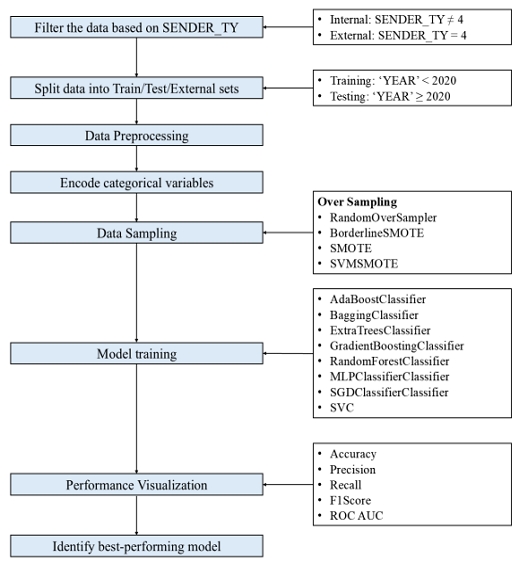
그림 2는 전체 시스템의 구성을 시각화 한 것이다. 순서는 크게 데이터 전처리와 모델 학습 및 성능 시각화로 나눠지며, 자세한 과정은 다음과 같다.
먼저 표 1은 논문에서 사용된 용어를 정의하는 표이다. 이는 ‘0600’ 그리고 ‘1300’이 무엇을 의미하는지 나타낸다. 표 2는 실험에서 사용된 변수들의 특징을 설명하는 표이다. 이 표는 사용된 변수들의 정보와 데이터 형식에 대한 정보를 제공하여준다.
머신 러닝을 수행하기 전, 먼저 사용할 데이터에 대한 전처리를 수행한다. 먼저, KAERS DB 데이터를 사용할 열을 선택한 뒤 결측치를 0으로 대체한다. 이후 ‘INGR_CD’ 변수를 기준으로 약물을 4개의 그룹(group1 ~ group4)으로 분류하고, 분류되지 않은 데이터는 제거한다. 약물 투여 시작일과 종료일을 날짜 형식으로 변환한 후, 두 날짜의 차이를 계산하여 약물 투여 기간을 의미하는 새로운 ‘DOSAGE PERIOD’ 열을 생성한다. 그 후 ‘WHOART ARRN’과 'WHOART SEQ’ 값을 매칭하여 새로운 ‘WHOART soc1’ 열을 생성한다. 추가적으로, 머신 러닝 수행을 위하여 ‘INGR CD’, ‘PTNT SEX’, ‘DOSAGE ROUTE ID’, 그리고 ‘EFFICACY MEDDRA ENG NM’ 등의 범주형 데이터를 숫자로 매핑한다. 이후에는 ‘SENDER TY’ 값이 4인 데이터를 외부 검증 데이터로 분리하고, 나머지 데이터를 내부 검증 데이터로 나눠 데이터셋을 생성한다. 머신 러닝 학습 시 사용할 데이터의 생성을 위하여 연도를 뜻하는 ‘YEAR’ 값에 따라 내부 데이터를 학습용 데이터(2018-2019)와 테스트용 데이터(2020-2022)로 분리한다. 이때, 특정 인체 내 장기 장애에 대해 분석하기 위해서 ‘WHOART soc1’ 값을 기준으로 특정 질병 코드에 해당하는 경우 1, 아닌 경우 0으로 설정하여 타겟 Label을 정의한다. 우리가 제안한 방법에서는 소화기 장애를 의미하는 ‘0600’과 신장 장애를 의미하는 ‘1300’의 질환 코드를 이용한다. 따라서 Label 1에 해당하는 값이 ‘1300’인 경우, 그리고 ‘0600’인 경우 총 두 가지에 대해서 머신러닝이 수행된다. 마지막으로, 타겟 변수(‘WHOART soc1’)를 제거한 후, Train, Internal validation, 그리고 External validation dataset을 구성하여 머신 러닝을 위한 준비를 마친다.
다음으로, 앞선 전처리 과정에서 획득한 Train, Internal validation, 그리고 External validation 데이터를 이용하여 머신 러닝을 수행한다. 이때 사용한 모델은 Ada Boost Classifier[10], Bagging Classifier [11], Extra Trees Classifier [12], Gradient Boosting Classifier [13], Random Forest Classifier [14], MLP Classifier [15], SGD Classifier [16], 그리고 SVC [17]의 8개의 분류기 모델이다. 학습을 수행할 때에 있어서 샘플링을 이용하여 불균형 데이터를 처리한다. 사용된 샘플링 기법은 Over Sampling이며 모델은 RandomOverSampler, BorderlineSMOTE, SMOTE, 그리고 SVMSMOTE의 4종이다. 이때, 샘플링 실행 여부와 그 횟수에 따라 모델 성능을 비교하기 위하여 조건을 설정한다. 첫 번째로는, 샘플링 수행 횟수에 따른 성능을 비교하기 위해 동일하게 RandomOverSampler 모델을 사용하여 샘플링을 수행하지 않은 건, 샘플링을 1회 수행한 건, 샘플링을 5회 수행한 건에 대한 결과를 비교한다. 두 번째로는, 샘플링 모델 종류에 따른 결과를 비교하기 위하여 앞서 언급한 4 종의 샘플링 모델을 이용하여 훈련 데이터를 샘플링한 뒤 모델의 예측 성능을 비교한다. 모델 성능의 비교분석은 Accuracy, Precision, Recall, F1 Score, ROC AUC의 평가 지표를 시각화하여 수행한다.
| (1) |
| (2) |
| (3) |
| (4) |
결과 분석에 사용된 평가 지표는 수식 (1)부터 (4)까지에 나와 있다. TP (True Positive)는 모델에서 실제 참인데 분류 모델 예측 결과 참으로 예측한 경우를, TN (True Negative)는 실제로 거짓인 경우를 정확히 거짓으로 예측한 경우를, FP (False Positive)는 실제로 거짓인데 참으로 예측한 경우를, FN (False Negative)는 실제로 True인데 예측은 거짓이라 판단한 경우를 뜻한다. 수식 (1)은 양수 및 음수 모델 예측에서 계산된 Accuracy 값을 설명한다. Accuracy가 높을수록 샘플을 분류하는 모델의 전반적인 성능이 더 우수하다는 것을 나타낸다. 수식 (2)는 분류 모델에 의한 양성 예측의 정확도를 정량화하는 지표인 Precision 값에 대해 설명하며, 이 값이 높을수록 오탐 예측이 적다는 것을 뜻한다. 수식 (3)은 Recall에 대해 설명하며, 이는 데이터 세트의 진양성 샘플 중 진양성 예측의 백분율로 계산된다. 이는 정탐률의 민감도를 나타내며, Recall이 높을수록 오탐 예측이 적다는 것을 나타낸다. 수식 (4)는 F1 score에 대한 식이며, 이는 Precision과 Recall로 계산된다. 이는 Precision과 Recall을 동시에 고려하는 분류 모델의 성능을 측정하는 척도를 제공한다. F1 점수가 높을수록 정확도와 회수율 모두에서 모델의 전반적인 성능이 더 우수하다는 것을 나타낸다.
Ⅳ. 실험 결과
4-1 Correlation Analysis Results
그림 3은 Label 1, 즉 ‘1300’ 및 ‘0600’의 신장 장애와 소화기 장애에 대한 데이터와 ‘DOSAGE_ROUTE_ID’, ‘INGR_CD’, ‘PTNT_OCCURSYMT_AGE’, ‘PTNT_SEX’, 그리고 ‘SENDER_TY’의 특징들에 대한 scattor plot을 시각화 한 그림이다. 그림 3에 (a)의 ‘DOSAGE_ROUTE_ID’ 산점도 그래프에서는 모든 데이터가 동일한 위치에 집중되어 있어, ‘DOSAGE_ROUTE_ID’ 변수에서의 값이 거의 동일하거나 제한된 범위 내에서만 분포하고 있음을 확인했다. 또한, 그림 3에 (b)의 ‘INGR_CD’의 산점도 그래프에서는 데이터가 산재하여 다른 변수 간의 관계가 명확하지 않거나, 데이터가 불균형하게 분포되어 있음을 확인했다. 그림 3에 (c)의 ‘PTNT_OCCURSYMT_AGE’의 산점도 그래프에서는 환자 발생 증상 연령의 분포가 제한적임을 확인했다. 또한, 그림 3에 (d)의 ‘PTNT_SEX’의 산점도 그래프에서는 성별에 따른 데이터 분포가 거의 균일하게 나타나며, 성별이 다른 변수와의 관계에서 큰 영향을 미치지 않음을 확인했다. 마지막으로, 그림 3에 (e)의 ‘SENDER_TY’의 산점도 그래프에서는 ‘SENDER_TY’ 변수에 따라 데이터가 일부 증가하는 경향을 보임을 확인했다.
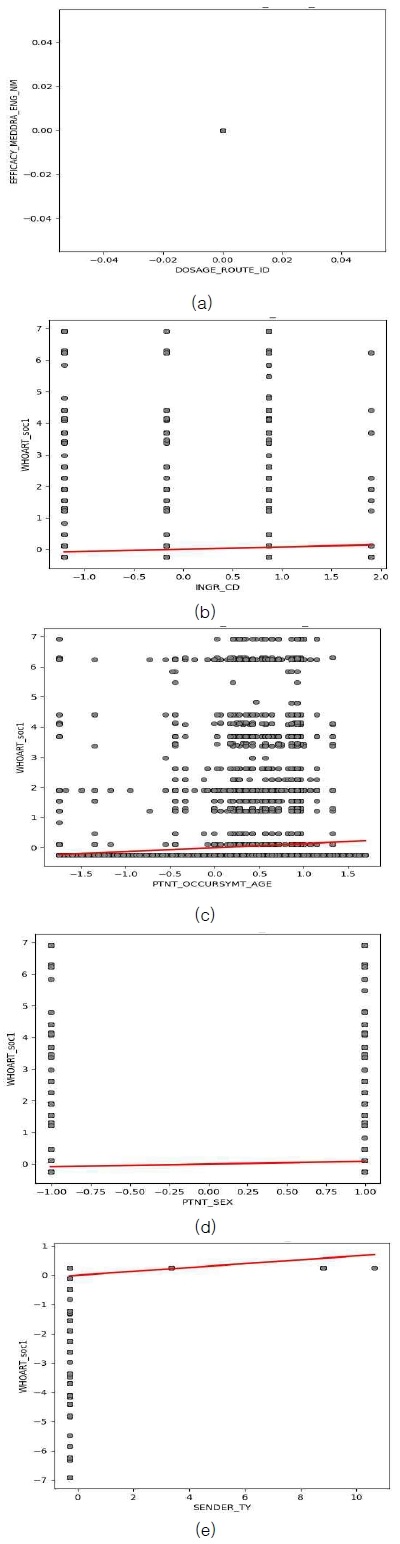
Visualization of CCA scatter plot for ‘1300’ and ‘0600’ data without sampling, (a) 'DOSAGE_ROUTE_ID', (b) 'INGR_CD', (c) 'PTNT_OCCURSYMT_AGE', (d) 'PTNT_SEX', and (e) 'SENDER_TY’
표 3은 Label 1에 대한 데이터와 각 특징들 간의 상관 분석을 수행한 결과를 나타낸다. Pearson correlation, Spearman correlation, Kendall correlation 총 3 종을 이용하여 ‘DOSAGE_ROUTE_ID’, ‘INGR_CD’, ‘PTNT_OCCURSYMT_AGE’, ‘PTNT_SEX’, 그리고 ‘SENDER_TY’의 특징들에 대한 상관 분석을 수행한 결과, 각 feature가 Label에 미치는 영향을 확인했다. ‘DOSAGE_ROUTE_ID’의 경우 상관계수와 p-value값이 nan으로 나타나며 결측치로 인해 상관 분석이 제대로 수행되지 않음을 확인했다. ‘INGR_CD’의 경우 Pearson, Spearman, 그리고 Kendall 상관 계수 모두에 대해서 상관관계가 거의 없는 것으로 나타났다. ‘PTNT_SEX’과 ‘SENDER_TY’에서도 역시 동일하게 상관 계수 값이 매우 작아 상관관계가 거의 없는 것으로 나타났다. ‘PTNT_OCCURSYMT_AGE’의 경우 선택된 5종의 특징들 중 가장 상관관계가 높은 것으로 확인되었다. Pearson 상관 분석을 통해 타겟 Label에 대해 약한 양적 선형 관계를 나타냄을 확인할 수 있었고, Spearman과 Kendall에서는 양의 비선형 관계를 갖고 있음을 확인했다. 이 값은 Label 1으로 설정된 ‘0600’과 ‘1300’의 신장 장애, 소화기 장애 데이터에 대해 모두 동일하게 나타났다.
4-2 Model Evaluation of Performance Metrics
표 4는 샘플링을 수행하지 않은 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애 및 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. ‘1300’의 경우 Accuracy는 대부분의 모델에서 매우 높은 값을 보였다. 하지만 F1-score, Precision, 그리고 Recall 값이 0에 가까운 경우가 많았다. AUC score 값은 0.5에 가까운 값부터 0.9 이상의 값을 보였다. ‘0600’ 역시 Accuracy는 대부분의 모델에서 매우 높은 값을 보이며, F1-score, Precision, 그리고 Recall 값이 작게 나타났다. 또한, AUC score 값 역시 ‘1300’과 같이 높은 값을 보였다.
표 5는 RandomOverSampler를 이용하여 샘플링을 1회 수행한 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애와 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. 두 표 모두 External validation 데이터에 대해서는 대부분의 모델에서 F1score, Precision, 그리고 Recall 값이 0에 가깝게 나타났으며 Accuracy 역시 낮게 나타났다. Internal validation 데이터에 대해서도 비슷한 양상을 보였으나, Train 데이터의 경우 대부분의 모델이 높은 성능을 보이는 것으로 나타났다.
표 6은 RandomOverSampler를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애와 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. Accuracy의 경우 External validation 및 Internal validation 데이터에서 낮게 나타났으며 F1-score, Precision, 그리고 Recall 등의 성능 지표 역시 낮게 나타났다. 반면 Train 데이터에서는 성능 지표 값이 높게 나타났다.
표 7은 BorderlineSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애와 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. 일부 모델에서 Train 데이터에 대해 높은 성능을 보이지만, External validation 및 Internal validation 데이터에 대해서는 성능이 낮게 나타났다.
표 8은 SMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애와 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. External validation, Internal validation, Train 데이터에서 보다 균형 잡힌 성능을 보이나 일부 모델의 경우 External validation, Internal validation 데이터에 대해서 성능 저하가 있는 것으로 나타났다.
표 9는 SVMSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’과 ‘0600’의 코드로 분류되는 신장 장애와 소화기 장애 데이터에 대한 모델 성능을 비교한 표이다. Train data에 대해서는 높은 성능을 나타내며 External validation, Internal validation 데이터에 대해서 성능이 더 개선되어 나타나나 여전히 성능 저하가 존재했다.
표 10은 표 4~9까지의 결과에 대한 평균값을 나타낸 표이다. 샘플링 수행 횟수를 기준으로 비교하였을 때, 샘플링을 전혀 적용하지 않았을 때의 성능 지표는 상당히 높은 Accuracy (0.975)를 기록했지만, F1 score (0.015), Precision (0.067), Recall (0.041) 등에서는 매우 낮은 값을 보였다. 샘플링을 1회 적용한 RandomOverSampler의 경우 Accuracy는 0.853으로 감소하였지만, F1 score (0.278), Precision (0.263), 그리고 Recall (0.450)에서 개선되는 모습을 보였다. 반면, 샘플링을 5회 적용했을 때, RandomOverSampler의 Accuracy는 0.721로 더욱 낮아졌고, F1 score (0.412)와 Recall (0.450)에서의 개선은 보였으나, Precision에서는 달라지는 값이 없었다. 따라서 샘플링을 1회 적용한 건과 5회 적용한 건 간의 평균적인 변화는 Accuracy에서 약 0.132의 감소가 있었고, F1 score에서는 약 0.134의 증가가 있었다. 샘플링 모델의 종류에 대해 성능을 비교했을 때, BorderlineSMOTE와 SVMSMOTE는 각각 F1 score (0.285 그리고 0.253)과 Recall (0.485 그리고 0.467)에서 상대적으로 높은 성능을 보였다.
4-3 ROC Curves of Prediction Model
그림 4(a)~(c)는 샘플링을 수행하지 않은 8종의 모델의 Label 1, 즉 ‘1300’의 코드로 분류되는 신장 장애 데이터에 대한 ROC curve를 표현한 그림이다. (a), (b), 그리고 (c)는 순서대로 Train, Internal validation, External validation 데이터를 구분한 것이다. External validation과 Internal validation 데이터에 대해서는 MLPClassifier가, Train에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다. 그림 4(d)~(f)는 샘플링을 수행하지 않은 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 소화기 장애 데이터에 대한 ROC curve를 표현한 그림이다. (d), (e), 그리고 (f)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation과 Internal validation 데이터에 대해서는 AdaBoostClassifier가, Train에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다.
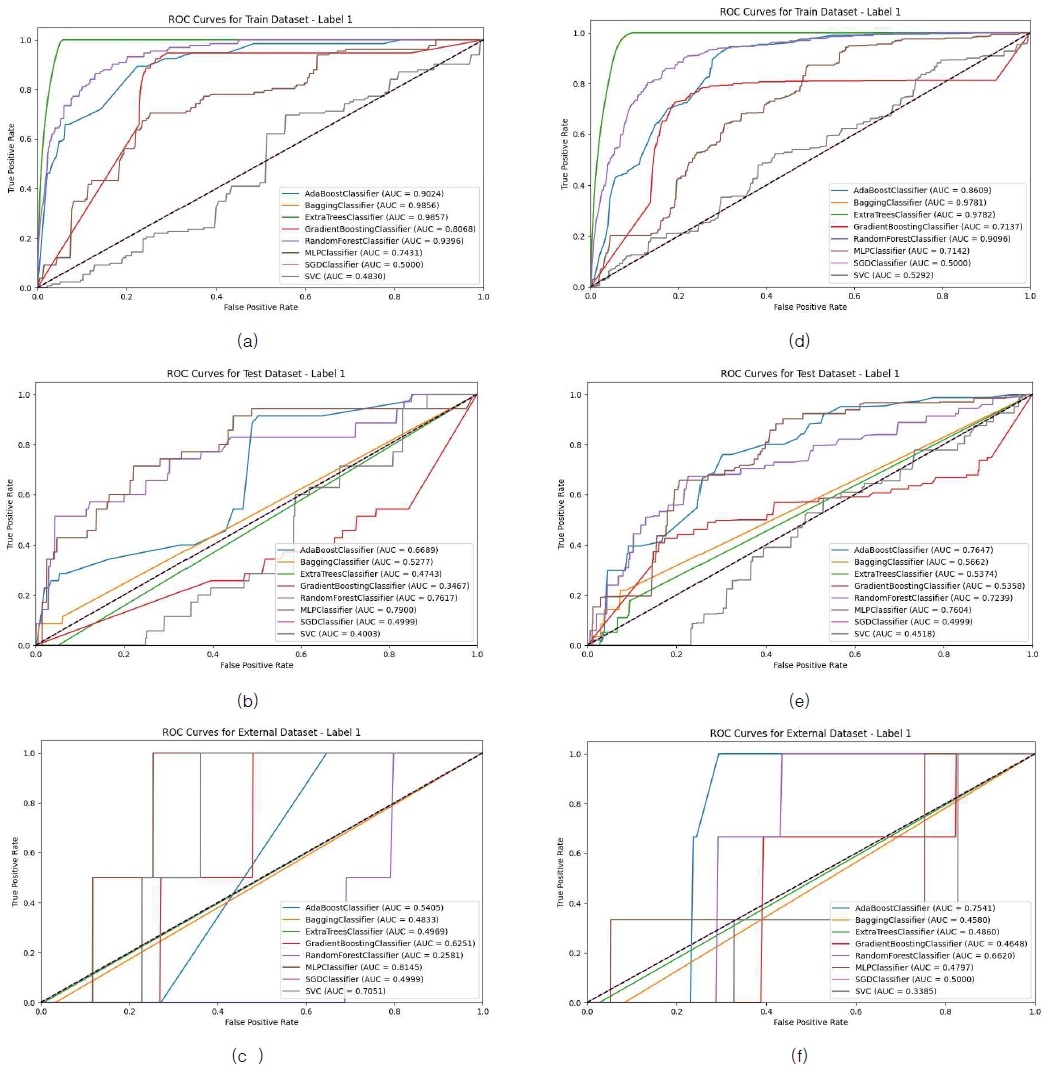
Visualization of ROC curves for Label 1 data without sampling, (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
그림 5(a)~(c)는 RandomOverSampler를 이용하여 샘플링을 1회 수행한 8종의 모델의 Label 1, 즉 ‘1300’의 코드로 분류되는 신장 장애 데이터에 대한 ROC curve를 표현한 그림이다. (a), (b), 그리고 (c)는 순서대로 Train, Internal validation, External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 SGDClassifier가, Internal validation 데이터에 대해서는 MLPClassifier가, Train에서는 BaggingClassifier, ExtraTreesClassifier, GradientBoostingClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다. 그림 5(d)~(f)는 RandomOverSampler를 이용하여 샘플링을 1회 수행한 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 소화기 장애 데이터에 대한 ROC curve를 표현한 그림이다. (d), (e), 그리고 (f)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 AdaBoostClassifier가, Internal validation 데이터에 대해서는 SVC가, Train에서는 BaggingClassifier, ExtraTreesClassifier, 그리고 GradientBoostingClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다.
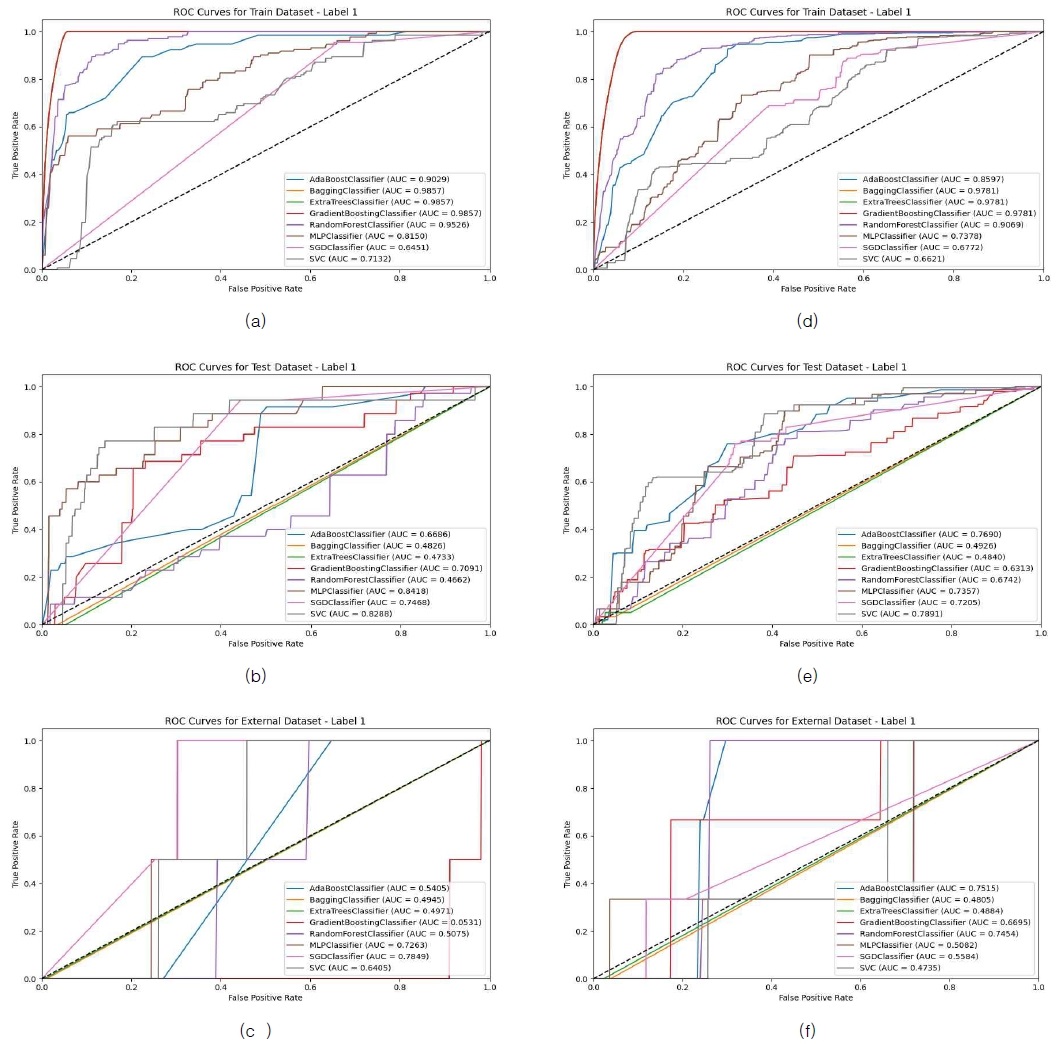
Visualization of ROC curves for ‘1300’ data aacross 1 iterations of RandomOverSampler sampling, (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
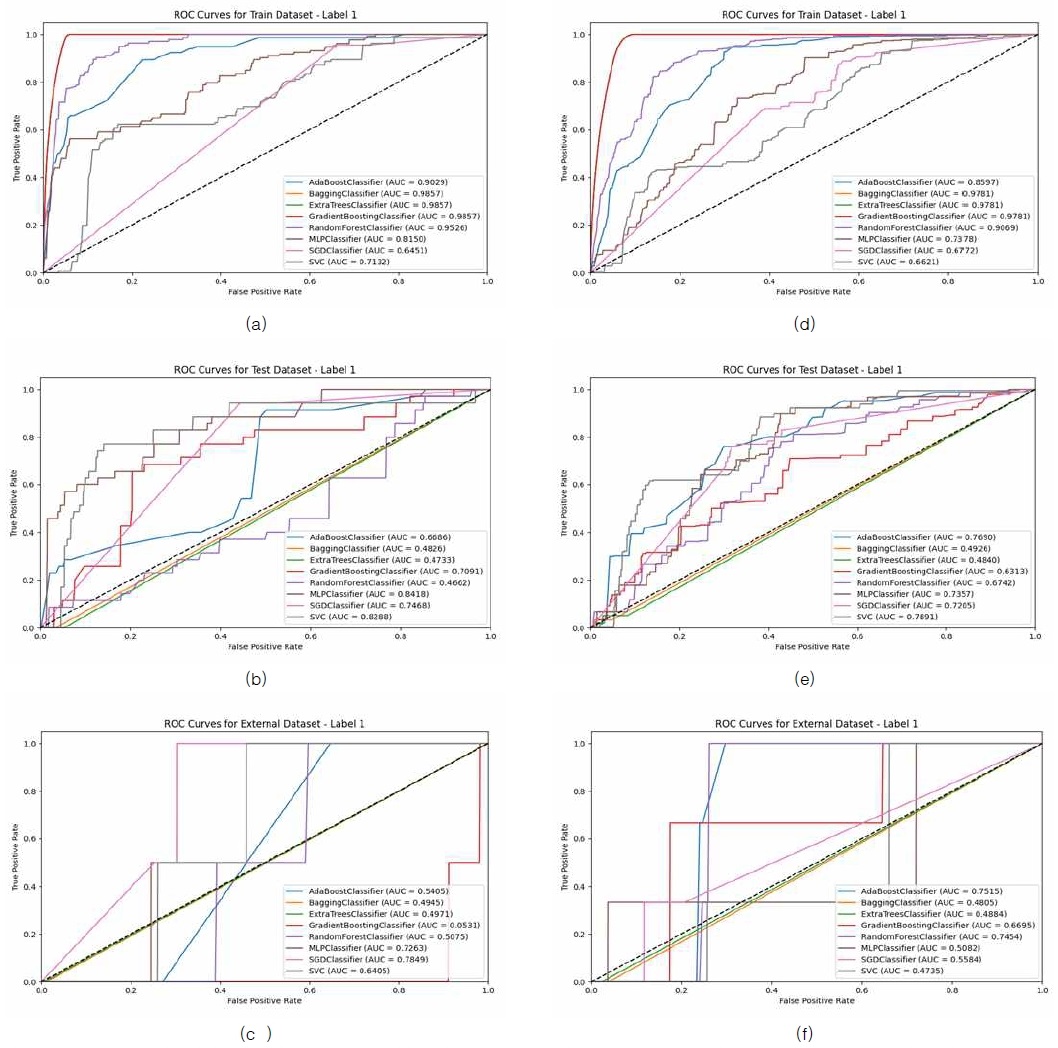
Visualization of ROC curves for ‘1300’ data aacross 5 iterations of RandomOverSampler sampling, (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
그림 7 (a)~(c)는 BorderlineSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’의 코드로 분류되는 신장 장애 데이터에 대한 ROC curve를 표현한 그림이다. (a), (b), 그리고 (c)는 순서대로 Train, Internal validation, External validation 데이터를 구분한 것이다. External validation과 Internal validation 데이터에 대해서는 SVC가, Train 데이터에서는 BaggingClassifier과 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다. 그림 7의 (d)~(f)는 BorderlineSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 소화기 장애 데이터에 대한 ROC curve를 표현한 그림이다. (d), (e), 그리고 (f)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 RandomForestClassifier가, Internal validation 데이터에 대해서는 SVC가, Train 데이터에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다.
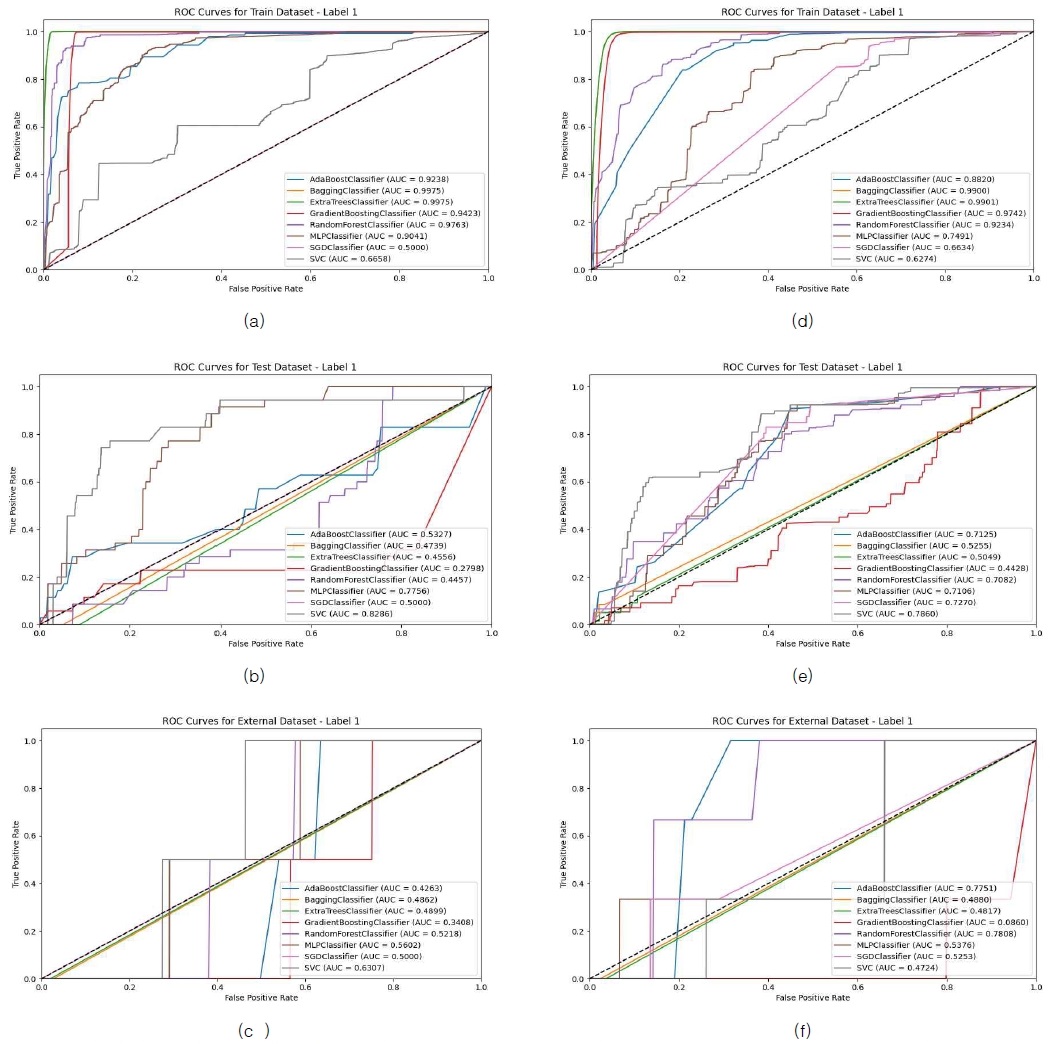
Visualization of ROC curves for ‘1300’ data across 5 iterations of BorderlineSMOTE sampling (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
그림 8 (a)~(c)는 SMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘1300’의 코드로 분류되는 신장 장애 데이터에 대한 ROC curve를 표현한 그림이다. (a), (b), 그리고 (c)는 순서대로 Train, Internal validation, External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 SGDClassifier가, Internal validation 데이터에 대해서는 MLPClassifier가, Train 데이터에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다. 그림 8의 (d)~(f)는 SMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 소화기 장애 데이터에 대한 ROC curve를 표현한 그림이다. (d), (e), 그리고 (f)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 AdaBoostClassifier가, Internal validation 데이터에 대해서는 SVC가, Train 데이터에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다.
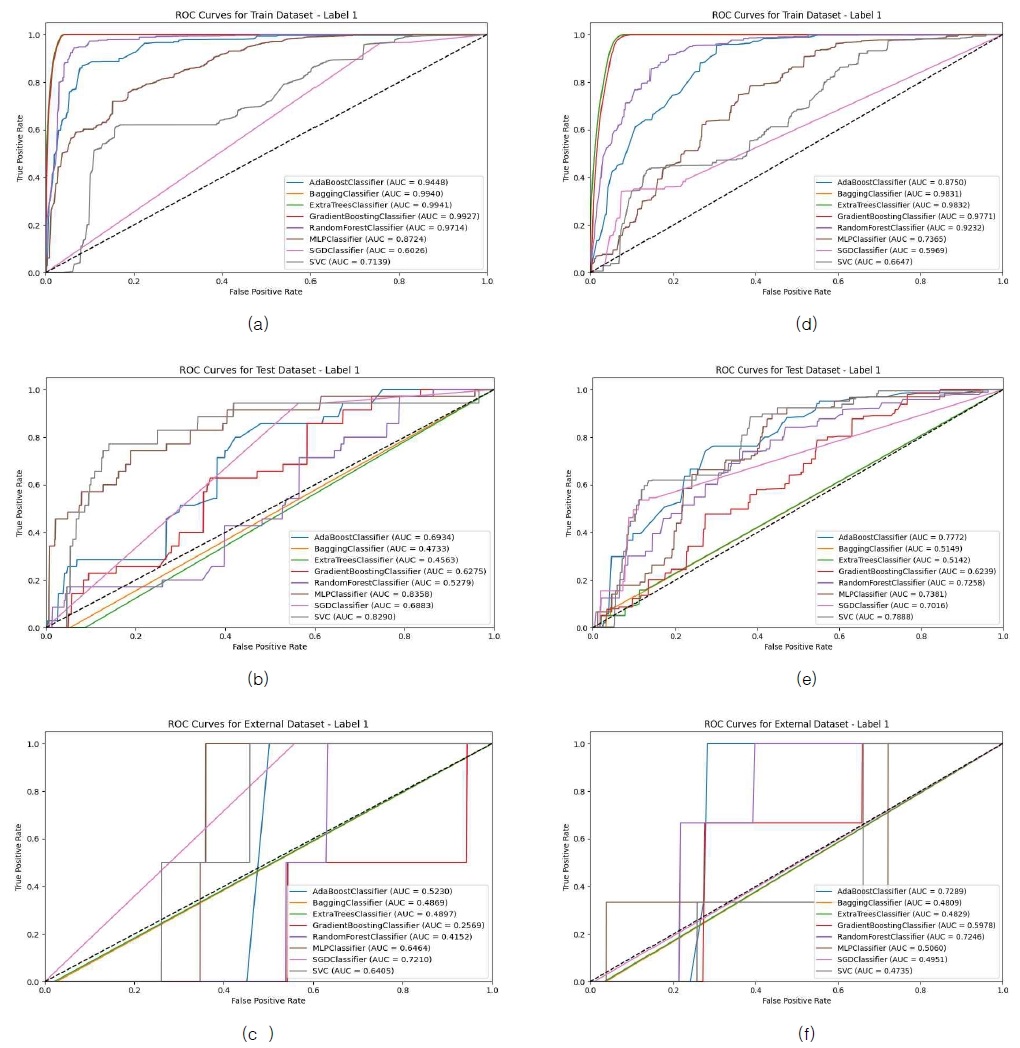
Visualization of ROC curves for ‘1300’ data across 5 iterations of SMOTE sampling (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
그림 9의 (a)~(c)는 SVMSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 신장 장애 데이터에 대한 ROC curve를 표현한 그림이다. (a), (b), 그리고 (c)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 GradientBoostingClassifier가, Internal validation 데이터에 대해서는 SVC가, Train 데이터에서는 BaggingClassifier와 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다. 그림 9의 (d)~(f)는 SVMSMOTE를 이용하여 샘플링을 5회 수행한 8종의 모델의 Label 1, 즉 ‘0600’의 코드로 분류되는 소화기 장애 데이터에 대한 ROC curve를 표현한 그림이다. (d), (e), 그리고 (f)는 순서대로 Train, Internal validation, 그리고 External validation 데이터를 구분한 것이다. External validation 데이터에 대해서는 AdaBoostClassifier가, Internal validation 데이터에 대해서는 SVC가, Train 데이터에서는 ExtraTreesClassifier가 모델 분류 성능이 가장 뛰어난 것으로 나타났다.
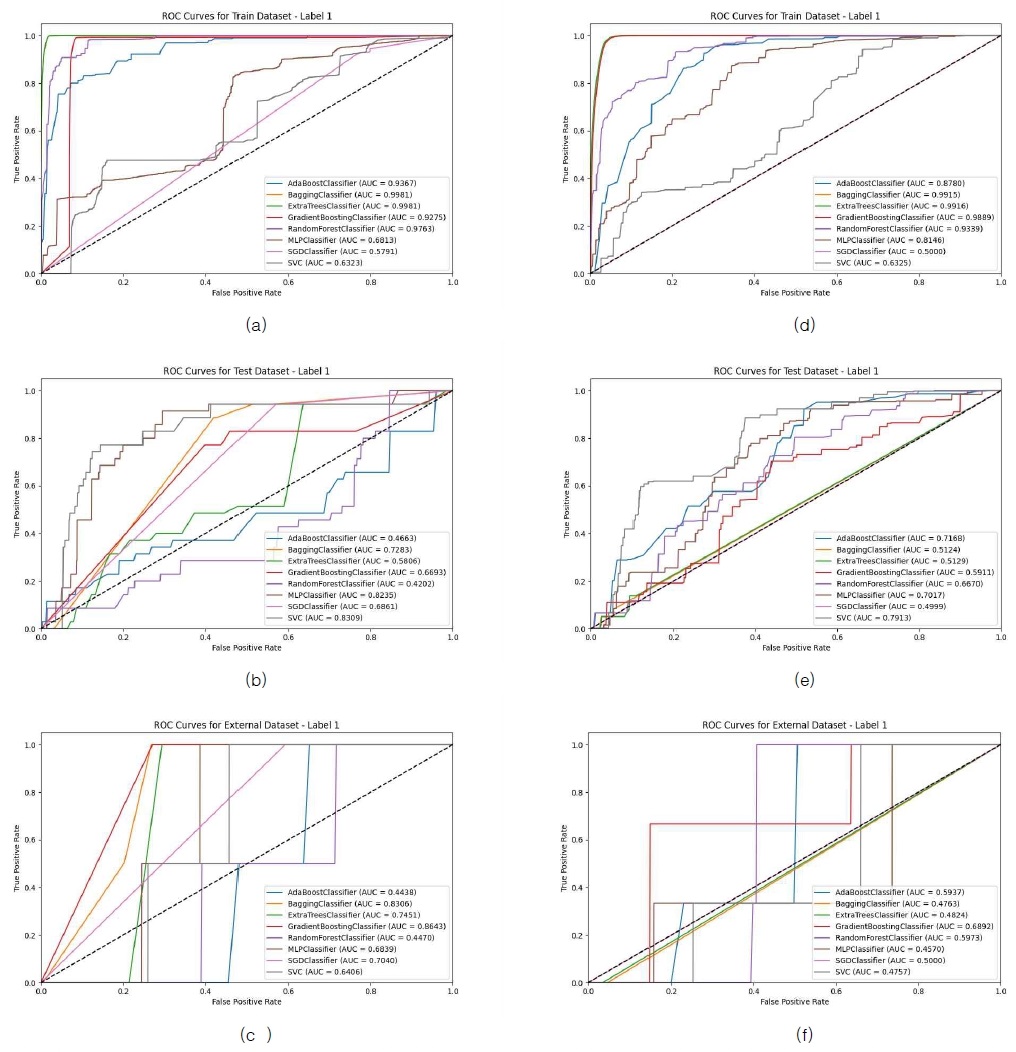
Visualization of ROC curves for ‘1300’ data across 5 iterations of SVMSMOTE sampling (a) Train dataset for ‘1300’, (b) Internal validation dataset for ‘1300’, (c) External validation dataset for ‘1300’, (d) Train dataset for ‘0600’, (e) Internal validation dataset for ‘0600’, and (f) External validation dataset for ‘0600’
이어서 샘플링 반복 횟수에 따른 모델 성능에 대해서 설명한다. 먼저, 샘플링을 수행하지 않은 건에 대해서는 Accuracy가 대부분의 모델에서 매우 높은 값을 보이고 있음을 확인할 수 있었지만, F1-score, Precision, Recall 값이 0에 가까운 값으로 나타났다. 이를 통해 대부분의 샘플이 0 (negative class)에 해당되는 등, 모델이 거의 모든 샘플을 0으로 예측하며 1 (positive class)에 대해 제대로 예측하지 못함을 확인했다. AUC score 역시 높은 값을 갖으며 모델이 불균형 클래스 간의 구분 능력을 어느 정도 가지고 있음을 나타냈지만, F1-score나 Recall이 매우 낮아 positive class (1)를 제대로 예측하지 못하고 있음을 재차 확인했다. 결국, 샘플링을 수행하기 전에는 불균형 데이터의 문제가 해결되지 않는다는 것을 알 수 있었다.
RandomOverSampler를 이용하여 샘플링을 1회 수행한 건에 대해서는 External validation과 Internal validation 데이터에 대해서 대부분의 모델에서 F1 score, Precision, 그리고 Recall 값이 0으로 나타나며 외부 데이터에 대해 모델이 타겟 라벨(1)을 분류하는 데 어려움을 겪고 있음을 확인했다. 표 4와 5의 성능 지표 값을 통해 이 경우 타겟 Label(1)를 잡아내긴 했지만, 다른 성능 지표가 매우 낮아 유의미한 분류를 하지 못하며 다수의 오탐(False Positive)이 존재함을 확인했다. 이 케이스 역시 대부분의 모델에서 Recall이 0이거나 매우 낮게 나타나며, 타겟 클래스 (1)를 거의 탐지하지 못한 것으로 나타났다. 또한, AUC score 값이 샘플링을 수행하지 않은 경우보다 일반적으로 낮아지며 클래스 구분 성능이 떨어졌음을 확인했다.
RandomOverSampler를 이용하여 샘플링을 5회 수행한 건에 대해서는 Internal validation 및 Train 성능에서 일부 성능 지표가 개선되었음을 확인했다. 하지만 External validation 데이터에 대해서는 여전히 F1score, Precision, Recall 값이 0에 가깝게 나타나는 것이 많았다. 따라서 모델이 외부 데이터에 대해 과적합되었거나, 불균형 문제가 해결되지 않았음을 추정할 수 있었다. Train 데이터의 경우 대부분의 모델들이 비교적 높은 성능 지표 값을 가졌으며, 이를 통해 샘플링 기법을 통해 균형 있는 학습 수행이 가능하다 판단했다. 더하여, AUC score 값이 샘플링을 1회 수행한 건 보다 개선된 경우가 많은 것으로 미루어 보았을 때, 클래스 구분 성능이 향상됨을 알 수 있었다.
표 9의 평균 결과에 따르면, 샘플링을 수행 하였을 때에 비해 수행하지 않았을 때에 F1 score, Precision, Recall 등에서는 매우 낮은 값을 보이며 반복 횟수를 늘려감에 따라 값이 개선됨을 확인했다. 이런 결과를 통해 데이터 불균형이 모델 학습에 영향을 미치고 있으며 샘플링 반복 횟수를 증가시킴에 따라 이런 불균형이 개선될 수 있을 것이라 사료된다.
본 연구는 샘플링 모델 유형에 따른 모델 성능에 대해서 설명한다. 첫째로, RandomOverSampler를 이용하여 샘플링을 5회 반복한 결과, 성능 지표들이 External validation 및 Internal validation 데이터에 대해서 낮게 나타났으며 Train 에 대해서는 더 높은 지표를 가졌다. BorderlineSMOTE를 이용한 건에 대해서는 RandomOverSampler와 마찬가지로, 성능 지표가 Train 데이터에 비해 External validation 및 Internal validation 데이터에서 더 낮게 나타났다. 특히, BorderlineSMOTE의 경우 RandomOverSampler와 비교했을 때 일부 모델에서 성능 향상을 보였다. SMOTE의 경우 데이터셋 전반에 걸쳐 균형 잡힌 성능을 보였으나, Internal validation 데이터의 성능 지표가 여전히 Train 데이터에 비해서 낮게 나타났다. SVMSMOTE의 경우 Internal validation 데이터에 대한 성능 지표가 BorderlineSMOTE 및 RandomOverSampler에 비해 향상된다는 특징을 가졌다. 하지만 Train 데이터에 대한 성능 지표가 역시 Internal validation 보다 높게 나타났다.
표 9의 평균 결과에 따르면, BorderlineSMOTE와 SVMSMOTE의 두 샘플링 모델이 데이터의 경계에 있는 샘플을 더 잘 다루어 불균형 문제를 개선하는 데 효과적임을 시사한다. 결론적으로, 샘플링 기법을 적용함으로써 데이터 불균형 문제를 어느 정도 해결할 수 있으며, 특히 BorderlineSMOTE와 SVMSMOTE가 상대적으로 더 나은 성능을 나타내어 효과적인 대안으로 평가된다. 하지만 대부분의 모델이 External validation 및 Internal validation 데이터 보다 Train 데이터에서 더 높은 성능을 보이는 것으로 보아 모델이 과적합(Over-fitting)되었을 가능성이 존재하는 것을 확인했다.
Ⅴ. 결 론
약물 이상 사례 예측에서 인체 내 장기의 장애 현상을 분석하는 것은 환자의 안전과 의료 비용 절감에 중요한 역할을 수행한다. 이에 균형하게 발생하는 약물 이상 사례를 다루기 위해 본 연구는 인체 내 장기의 장애 현상 분석을 위한 새로운 머신러닝 접근법을 제안하였다. 특히, 데이터 불균형 문제를 해결하기 위하여 샘플링 횟수와 모델의 종류에 따라 제안한 모델의 성능을 비교하였다.
샘플링을 수행하지 않은 경우 모델의 Accuracy는 높았으나 F1-score, Precision, Recall 등의 지표가 매우 낮아 모델이 Label 1(positive class)을 제대로 예측하지 못하는 문제가 있음을 확인하였다. 이어, RandomOverSampler를 5회 반복 수행한 결과, Train 데이터에서는 1회 반복 건에 비하여 성능 평가 지표가 개선됨을 확인했다. 또한, 모델 종류에 따른 결과에 의하면 BorderlineSMOTE와 SMOTE를 사용한 경우 RandomOverSampler보다 성능이 높게 나타났으며, SVMSMOTE는 Internal validation 데이터에서 특히 우수한 성능을 보임을 확인하였다. 다만 대부분의 모델이 External validation 및 Internal validation 데이터보다 Train 데이터에서 높은 성능을 보이며 제안한 모델에게 과적합(over-fitting)의 가능성이 존재하는 것으로 나타났다. 하지만, 결론적으로 4종의 샘플링 모델을 적용한 결과 각 모델마다 다른 성능을 보였으며, 샘플링 모델의 반복 사용을 통해 불균형 데이터 문제를 완화하고 모델의 예측 성능을 향상시킬 수 있음을 확인했다.
향후 연구에서는 샘플링 기법을 성능에 맞게끔 함께 적용하고, 데이터의 품질과 양을 개선하는 등 모델의 일반화 성능을 높일 수 있는 방법을 제안하는 것이 필요할 것으로 사료된다. 추가적으로, 다양한 샘플링 기법을 조합한 앙상블 방법론을 탐색하여 각 기법의 장점을 극대화하는 방안을 모색하거나, 모델의 과적합 문제를 해결하기 위해 교차 검증 기법을 활용하고, 하이퍼파라미터 튜닝을 통해 모델의 최적화를 시도하는 등의 방법론을 적용할 수 있을 것이다.
References
-
J.-L. Montastruc, A. Sommet, I. Lacroix, P. Olivier, G. Durrieu, C. Damase-Michel, ... and H. Bagheri, “Pharmacovigilance for Evaluating Adverse Drug Reactions: Value, Organization, and Methods,” Joint Bone Spine, Vol. 73, No. 6, pp. 629-632, December 2006.
[https://doi.org/10.1016/j.jbspin.2006.09.002]

-
R. W. L. Leong and F. K. L. Chan, “Drug-Induced Side Effects Affecting the Gastrointestinal Tract,” Expert Opinion on Drug Safety, Vol. 5, No. 4, pp. 585-592, 2006.
[https://doi.org/10.1517/14740338.5.4.585]

-
L. M. Taft, R. S. Evans, C. R. Shyu, M. J. Egger, N. Chawla, J. A. Mitchell, ... and M. Varner, “Countering Imbalanced Datasets to Improve Adverse Drug Event Predictive Models in Labor and Delivery,” Journal of Biomedical Informatics, Vol. 42, No. 2, pp. 356-364, April 2009.
[https://doi.org/10.1016/j.jbi.2008.09.001]

-
P. Bate, P. Mendel, and G. Robert, Organizing for Quality: The Improvement Journeys of Leading Hospitals in Europe and the United States, Abingdon, UK: Radcliffe Publishing, 2008.
[https://doi.org/10.1201/b20730]

-
D. Yoon, E. K. Ahn, M. Y. Park, S. Y. Cho, P. Ryan, M. J. Schuemie, ... and R. W. Park, “Conversion and Data Quality Assessment of Electronic Health Record Data at a Korean Tertiary Teaching Hospital to a Common Data Model for Distributed Network Research,” Healthcare Informatics Research, Vol. 22, No. 1, pp. 54-58, January 2016.
[https://doi.org/10.4258/hir.2016.22.1.54]

-
R. Liu, M. D. M. AbdulHameed, K. Kumar, X. Yu, A. Wallqvist, and J. Reifman, “Data-Driven Prediction of Adverse Drug Reactions Induced by Drug-Drug Interactions,” BMC Pharmacology and Toxicology, Vol. 18, 44, June 2017.
[https://doi.org/10.1186/s40360-017-0153-6]

-
J. H. G. Scholl, F. P. A. M. van Hunsel, E. Hak, and E. P. van Puijenbroek, “A Prediction Model-based Algorithm for Computer-Assisted Database Screening of Adverse Drug Reactions in the Netherlands,” Pharmacoepidemiology & Drug Safety, Vol. 27, No. 2, pp. 199-205, February 2018.
[https://doi.org/10.1002/pds.4364]

-
R. Miotto, L. Li, B. A. Kidd, and J. T. Dudley, “Deep Patient: An Unsupervised Representation to Predict the Future of Patients from the Electronic Health Records,” Scientific Reports, Vol. 6, 26094, May 2016.
[https://doi.org/10.1038/srep26094]

-
R. Qureshi, M. Irfan, T. M. Gondal, S. Khan, J. Wu, M. U. Hadi, ... and T. Alam, “AI in Drug Discovery and Its Clinical Relevance,” Heliyon, Vol. 9, No. 7, e17575, July 2023.
[https://doi.org/10.1016/j.heliyon.2023.e17575]

- Scikit-Learn. Ada Boost Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.ensemble.AdaBoostClassifier.html, .
- Scikit-Learn. Bagging Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.ensemble.BaggingClassifier.html, .
- Scikit-Learn. Extra Trees Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.ensemble.ExtraTreesClassifier.html, .
- Scikit-Learn. Gradient Boosting Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.ensemble.GradientBoostingClassifier.html, .
- Scikit-Learn. Random Forest Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.ensemble.RandomForestClassifier.html, .
- Scikit-Learn. MLP Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.neural_network.MLPClassifier.html, .
- Scikit-Learn. SGD Classifier [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.linear_model.SGDClassifier.html, .
- Scikit-Learn. SVC [Internet]. Available: https://scikit-learn.org/stable/modules/generated/sklearn.svm.SVC.html, .
저자소개

2021년~2024년: 이화여자대학교 (공학사-휴먼기계바이오공학부)
※관심분야:의료머신러닝, 생체의공학, 의료메카트로닉스 등

2009년:경희대학교 대학원 (의과학석사)
2010년:플로리다 주립대학 (팜디) / University of Florida (Pharm.D.)
2013년~2014년: 삼성서울병원
2014년~2015년: 서울대학교병원
2015년~2019년: 한국의약품안전관리원
2021년~현 재: 동덕여자대학교 약학대학 조교수
※관심분야:약물치료학, 항생제 안전성 및 유효성, 항암제 안전성 및 유효성

2020년:한성대학교 (공학석사)
2022년~2023년: 광운대학교 초빙교수
2023년~현 재: 한성대학교 기초교양학부 조교수
※관심분야:의료 딥러닝