메시지 패싱 그래프 기반 딥러닝 모델을 활용한 화합물의 심장독성 예측
Copyright ⓒ 2024 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
hERG 채널은 심장의 전기 활동에 필수적이며, 이 채널을 차단하는 물질은 심각한 심장 독성 효과를 일으킬 수 있다. 인실리코 예측 모델은 hERG 차단제를 효율적으로 선별할 수 있어 시간과 자원을 절약할 수 있다. 이전 접근법은 예측 결과를 해석하고 분자 구조-기능 관계를 이해하는 데 어렵다. 본 연구에서는 공개 데이터베이스로부터 화합물을 수집하여 12,920개의 데이터셋을 구축하였다. 화합물의 그래프 구조를 고려하는 그래프 신경망(GNN) 가운데 메시지 패싱 신경망(MPNN)을 활용하여 특징 벡터를 추출하고, 이를 구조적ㆍ물리화학적 특성과 결합하여 최종 hERG 차단제를 예측하였다. 해당 모델은 AUROC는 0.864 (±0.009), AUPR은 0.907 (±0.010)의 성능을 달성하였다. 실험 결과, 제안된 모델은 그래프 특징 벡터를 통합하여 분자 특성을 효과적으로 반영하고 분자 간의 관계를 예측하여 hERG 차단제를 예측할 수 있음을 시사한다. 본 연구는 약물 개발과정에서 예비 도구로 활용되어 심장독성을 조기에 평가할 수 있을 것이다.
Abstract
The hERG channel plays a critical role in the heart's electrical activity, and compounds that block this channel can lead to severe cardiotoxicity. In silico prediction models can efficiently screen for hERG blockers, saving time and resources. However, previous approaches find it difficult to interpret predictions and understand molecular structure–function relationships. In this study, we collected compounds from public databases to construct a dataset of 12,920 compounds. We introduced a GNN (graph neural network) to predict hERG blockers by analyzing the graph structures of compounds. We employed a MPNN (message passing neural network) to extract feature vectors from these molecular graphs, combining them with structural and physicochemical properties to improve prediction accuracy. Our model achieved an AUROC of 0.864 (±0.009) and an AUPR of 0.907 (±0.010). Experimental results show that the proposed model effectively captures molecular characteristics through graph feature vectors and accurately predicts the relationships between molecules, identifying hERG blockers. This model can serve as an early-stage tool in drug development, enabling early cardiotoxicity evaluation.
Keywords:
Drug Toxicity, hERG Channel Blocker, Cardiotoxicity, Deep Learning, Graph Neural Network키워드:
약물 독성, hERG 채널 차단제, 심장 독성, 딥러닝, 그래프 신경망 네트워크Ⅰ. 서 론
인간 에테르-아-고-고 관련 유전자(hERG; human ether-a-go-go-related gene)가 암호화되는 hERG 채널은 심장 리듬을 조절하는 중요한 이온 채널이다. hERG 채널 차단제는 이러한 hERG 채널의 기능을 억제하는 화합물을 의미하며, 이는 잠재적으로 심장독성을 유발할 수 있다. 특정 리간드에 의해 hERG 채널이 차단되면 실신으로 이어질 수 있는 QT 증후군이나 부정맥을 유발할 수 있다[1],[2]. 이는 심전도에서 QT 간격이 길어지는 것으로 입증되는데, QT 간격은 심전도에서 Q 파가 시작되어 T 파가 끝날 때까지의 시간을 나타내며 심장의 재분극을 반영하는 중요한 지표이다[3],[4]. 세르틴돌과 같은 약물은 심전도에서 보이는 것처럼 QT 간격이 연장되는 치명적인 현상으로 인해 시장에서 퇴출당하였다. 따라서 특정 약물은 잠재적으로 심장 질환과 관련이 있을 수 있으므로 약물의 독성 평가는 개발 단계에서 매우 중요하다[5]. 이러한 단계는 신약의 안전성을 보장하는 데 필수적인 hERG 채널을 차단하는 약물의 위험성을 평가하기 때문에 중요하다.
방사성 리간드 결합 분석 및 QTc 분석과 같은 다양한 시험관 내 분석법이 개발되어 화합물의 hERG 채널에 대한 억제 효과를 평가하고 있다[6]. 그러나 세포의 구성 요소 간의 상호 작용을 분석하는 시험관 내 실험법은 시간과 비용이 많이 들어 비효율적일 수 있다[7]. 이런 한계를 극복하기 위해 많은 인실리코 방법이 제안되었다. 이 접근법은 화합물의 심장 독성 잠재력을 효율적이고 신속하게 평가하여 시간과 자원을 절약할 수 있다.
연구자들은 RF (random forest) 및 SVM (support vector machine)과 같은 기계학습 알고리즘을 사용하여 hERG 채널 차단 활성에 따라 화합물을 분류하였다[8],[9]. 또한 화합물의 fingerprint 기반 KNN (K-nearest neighbor) 알고리즘도 hERG 채널 차단을 예측하는 데 사용되었다[10]. 이러한 접근법을 통해 분자 특성을 활용하여 hERG 채널 차단제를 예측하는 예측 모델 개발에 상당한 노력이 투자되었다. 한 연구에서는 분자 구조에서 추출한 ECFP (extended connectivity fingerprint)를 활용하여 SVM을 통해 hERG 차단제를 예측한다[11]. ECFP는 분자의 구조적 특징을 반영하는 fingerprint로, 중심 원자를 기준으로 반지름에 따라 이웃 원자들의 연결성을 확장하여 표현한다[12]. 특히, hERG-att는 ECFP를 활용하여 self-attention과 FCL (fully connected layer) 신경망을 통해 hERG 차단제를 예측한다. hERG 차단제를 예측하는 모델은 일반적으로 ECFP와 같은 분자 지문을 통해 구조적 특징을 추출하는 데 의존한다. 최근에는 그래프 기반 구조를 활용하는 딥러닝 접근법으로 GNN (graph neural network), GCN (graph convolutional network), GAT (graph attention network) 등이 주목받고 있다[13]-[16].
본 연구에서는 메시지 패싱 그래프 신경망에서 추출한 특성을 결합하여 hERG 채널 차단제를 예측한다. 이 구조에서 엔티티는 노드로 표현되고 관계는 에지로 표현된다. GNN을 통해 각 분자의 고유한 구조적 특성을 반영한 벡터를 얻을 수 있으며, 이는 분자 간의 유사성 분석 및 예측 모델링에 유용하게 사용될 수 있다. 특히, 본 연구에서는 GNN 방법 중 MPNN (message passing neural network)을 사용하였다[17]. MPNN은 노드와 이웃 노드 간의 정보를 교환하는 메시지 전달 방식을 통해 노드 상태를 업데이트하는 방식으로, 이웃 노드 간의 단순 평균이나 가중합을 사용하여 정보를 집계하는 기존 분자 그래프 모델보다 특성을 잘 반영할 수 있다는 구조적 이점을 가진다. 노드 간의 관계(에지)뿐만 아니라 이웃 노드들의 특성까지 결합하기 때문에 그래프의 전체적인 구조적 패턴을 더 깊이 있게 학습할 수 있다. 이런 방식으로 MPNN은 그래프 구조에서 중요한 정보를 학습하며, 분자의 구조적 특성과 특징을 모두 반영한 벡터를 얻어 hERG 차단제를 예측한다.
또한 GNN에서 추출한 그래프 특징뿐만 아니라 분자의 구조적 특징과 물리화학적 특징도 함께 사용하여 예측 성능을 향상시켰다. 이를 통해 hERG 채널 차단제 예측 모델이 단순히 그래프 구조만이 아닌, 분자의 다양한 물리화학적 특성까지 고려하여 보다 정확한 예측을 할 수 있게 한다.
Ⅱ. 본 론
2-1 실험 데이터 수집 및 전처리
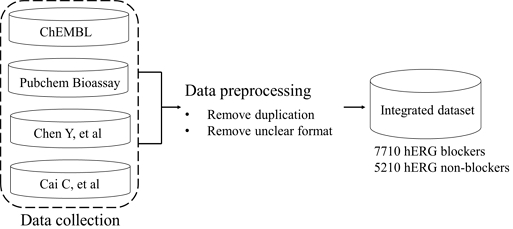
실험 데이터는 심장 독성과 관련된 데이터를 수집하기 위해 ChEMBL, PubChem Bioassay와 같은 공개 데이터베이스와 데이터의 다양성을 위해 외부 논문에서 보고된 데이터를 통합하여 구성된 통합 DB를 구축하였다[9],[18]-[20]. 해당 데이터베이스에는 화합물의 생물학적 활성을 나타내는 최대 억제 농도(IC50; half-maximal inhibitory concentration) 값을 포함하는 데이터를 수집하였다. IC50값은 생물학적 과정을 50% 감소시키는 데 필요한 농도를 의미하며, 10 μM 이상인 화합물은 hERG 비차단제로 분류하며, 10 μM 미만인 화합물은 hERG 차단제로 분류한다[21].
각 화합물은 ASCII 문자열을 사용하여 원자, 화학결합, 고리와 같은 성질을 표현하기 위해 SMILES (simplified molecular-input line-entry system)를 사용한다[22]. 동일한 분자 구조에 대해 다른 방식으로 작성된 SMILES 표현을 가질 수 있기 때문에 모호성을 나타낼 수 있다. 이를 위해 RDKit 라이브러리를 활용하여 사용하여 표준화된 canonical SMILES로 변환하였다[23]. 수집한 데이터 세트에서 중복된 화합물을 제거하였으며 불명확한 구조 형식을 제거하여 최종 12,920개의 고유 데이터 세트를 구성하였다. 데이터 수집 및 전처리 과정은 그림 1과 같다.
총 12,920개의 고유 화합물 데이터셋은 7,710개의 hERG 차단제와 5,210개의 hERG 바차단제로 구성되어 있다. 데이터셋은 훈련 세트(80%), 검증 세트(10%), 테스트 세트(10%)의 세 가지 하위 집합으로 나뉜다. 훈련 세트는 hERG 차단제가 6,168개이며 hERG 비차단제는 4,168개이다. 검증 세트와 테스트 세트에서는 hERG 차단제와 hERG 비차단제의 개수는 각각 771, 521개이다.
2-2 방법
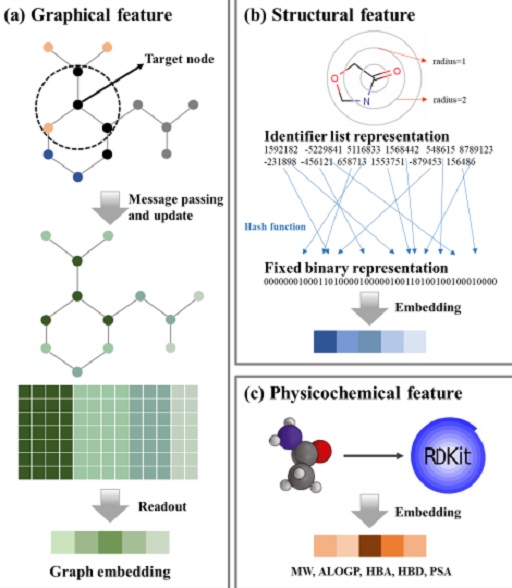
특징 추출은 크게 3단계인 그래프 특징 추출, 구조적 특징 추출, 물리화학적 특징 추출로 나뉜다. 그래프 특징에서는 분자 그래프 특징을 이용하여 분자의 고유한 특성을 파악하며, 구조적 특징에서는 화합물의 구조-활성 관계를 이진 벡터로 표현하여 추출한다. 물리화학적 특징은 5가지의 특성을 이용하여 추출한다. 해당 과정은 그림 2와 같다.
• 그래프 특징 추출
분자를 그래픽으로 표현하기 위해 RDkit 라이브러리를 사용하여 원자와 결합 특징을 얻었다. 각 원자 특징에는 10개의 특징이 있으며 원자 간의 연결을 나타내는 결합 특징은 4개의 특징으로 구성된다. 라디칼 전자와 같은 특징은 정수로 표현되며, 나머지 특징은 원핫 인코딩을 통해 표현된다. 원자 특징은 39-bit 벡터로, 결합 특징은 10-bit 벡터로 구성된다. 원자의 특성을 모델이 이해할 수 있는 형식으로 변환하기 위해 노드에 연결된 이웃 노드는 자신의 특징 벡터와 결합 특징 벡터를 결합하여 노드 정보를 초기화한다. 이 과정을 통해 각 원자의 이웃 노드는 임베딩 과정을 거쳐 자신의 특징 벡터와 결합 특징 벡터를 모두 결합한 특징 벡터로 변환된다.
• 구조적 특징 추출
화합물의 구조적 특징을 추출하기 위해 직경 3의 ECFP를 사용하였다. 이 원형 지문은 화합물의 화학적 특성을 벡터 형태로 인코딩하여 화합물의 구조-활성 관계를 모델링하고, 이를 모델에서 사용할 수 있는 이진형식으로 변환한다. 이 벡터는 화합물의 화학적 패턴의 유무를 나타내며, 모델이 생물학적 활성을 예측하는 데 중요한 역할을 한다.
• 물리화학적 특징 추출
화합물의 물리화학적 특성은 특정 기능을 설명하여 화합물의 대사, 배설 및 반응성을 결정하는 데 중요하다. 신약 개발의 단계에서는 이러한 물리화학적 특성을 활용한 평가가 신약 후보 물질의 가능성을 높이는 데 필수적이다[24]. 본 연구에서는 옥탄올-물 분할 계수(ALOGP; octanol-water partition coefficient), 분자량(MW; molecular weight), 위상학적 극성 표면적(TPSA; topological polar surface area), 수소 결합 수용체(HBA; hydrogen bond acceptors), 수소 결합 공여체(HBD; hydrogen bond donors)의 물리화학적 특징을 사용하였다. 한 연구에서는 ALOGP, MW, HBA, HBD, TPSA와 같은 물리화학적 특성과 hERG 채널의 억제 사이의 관계를 분석하였다[25]. 이 분석은 해당 특성이 hERG 채널과 어떻게 상호작용을 하는지에 대한 인사이트를 제공하며, 물리화학적 특성이 hERG 채널 차단제를 예측하는 데 중요한 역할을 한다는 것을 보여준다.
GNN은 그래프 구조 데이터를 효율적으로 처리하는 알고리즘으로, 생물학적 단백질 상호작용 네트워크, 분자 그래프 구조 등 다양한 분야에 적용되고 있다. 본 연구에서는 분자 그래프의 특징을 추출하기 위해 MPNN을 사용하였다. MPNN은 각 노드가 자신의 이웃 노드의 정보를 받아들여 특징 벡터를 업데이트하는 방식으로 동작한다. 이를 통해 분자의 구조적 특징을 효과적으로 학습할 수 있다. MPNN은 크게 두 가지 단계인 ‘Aggregate’와 ‘Update’로 구성된다.
‘Aggregate’ 단계에서는 각 노드의 이웃 노드로부터 메시지를 수신하여 이를 집계한다. 본 연구에서는 이웃 노드의 메시지를 인접 행렬을 사용하여 집계하였다. 이를 위해 각 노드의 초기 특성 벡터를 인접 행렬과 곱하여 이웃 노드의 정보를 반영한 집계 벡터를 생성하였다. 는 k번째 계층에서 노드 i의 집계 벡터, N(i)는 노드 i의 이웃 노드 집합, 는 이전 계층에서의 노드 j의 특징 벡터를 의미한다.
| (1) |
‘Update’ 단계에서는 집계된 벡터를 이용하여 각 노드의 특징 벡터를 업데이트한다. 노드의 특징 벡터는 집계 벡터를 더한 후 ReLU 활성화 함수를 거쳐 업데이트된 특징 벡터 를 생성한다. 여기서 W(k)는 k번째 가중치 행렬로, 학습 중에 각 계층에서 학습되는 파라미터이다.
| (2) |
각 노드의 특징 벡터를 업데이트한 후, 각 분자의 전체 특징 벡터를 생성하기 위해 그래프 내의 모든 노드 V의 특징 벡터를 합산하여 분자의 특징 벡터인 hmol를 생성하였다.
| (3) |
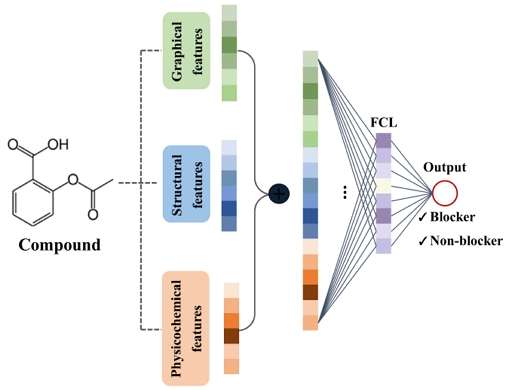
이를 통해 분자의 그래프 구조로부터 고유한 구조적 특성을 반영한 특징 벡터를 얻었으며, 해당 특징 벡터를 구조적ㆍ물리화학적 특성과 결합하여 최종 특성 벡터를 얻는다. 이후 예측을 위해 결합한 특성 벡터를 입력으로 가지는 FCL을 통해 hERG 차단제를 예측한다(그림 3).
2-4 평가 방법
모델의 예측 성능을 평가하기 위해 정확도, 재현율, 정밀도, 그리고 F1 점수를 이용하였다.
| (4) |
TN은 true negative로, 실제 음성인 데이터를 음성으로 예측한 경우를, TP는 true positive로, 실제 양성인 데이터를 모델이 양성으로 예측한 경우를 말한다. FN은 실제로는 양성이지만 모델이 잘못 예측하여 음성으로 판단한 경우를, FP는 실제로는 음성이지만 모델이 양성으로 잘못 예측한 경우이다. 해당 성능 지표를 활용하여 AUROC (area under the receiver operating characteristics)와 AUPR (precision-recall curve)을 사용하여 모델의 성능을 더욱 상세히 분석하였다. AUROC는 수신자 조작 특성 곡선 아래 면적을 나타내며, AUPR은 정밀도 재현율 곡선 아래 면적을 나타낸다. 이 값은 0에서 1까지의 범위를 가지며, 1에 가까운 값일수록 모델의 전반적인 예측 성능이 뛰어남을 의미한다.
K-fold 교차검증은 데이터를 K개의 균등한 fold로 나눈 후, 각 fold를 테스트로 이용하고 나머지 fold는 학습에 사용하는 방식이다. 이 과정이 k번 반복하여, k개의 서로 다른 모델 성능을 평가함으로써 데이터 분할에 의한 편차를 줄일 수 있다[26]. K-fold 교차검증의 장점을 유지하며, 데이터 불균형 문제를 해결하기 위해 본 연구에서는 stratified k-fold 교차검증을 사용하였다. 연구에서 사용된 hERG 차단제와 비 차단제의 비율은 1.48:1로, 극단적으로 불균형한 데이터는 아니지만, 모델이 다수 클래스에 편향된 예측을 하여 과대 적합이 발생할 수 있다. 특히 일반적인 k-fold에서는 모델 학습에 사용된 데이터와 성능 평가에 사용된 데이터의 클래스 분포가 달라질 수 있어, 모델 성능 평가에 영향을 줄 수 있다. 그러나, stratified k-fold는 각 fold에서 클래스 비율을 유지하도록 나누기 때문에 성능 평가가 일관성 있게 이루어지고, 모든 클래스에 대한 균형 잡힌 평가가 가능하다[27],[28].
Ⅲ. 실험결과
3-1 성능 평가
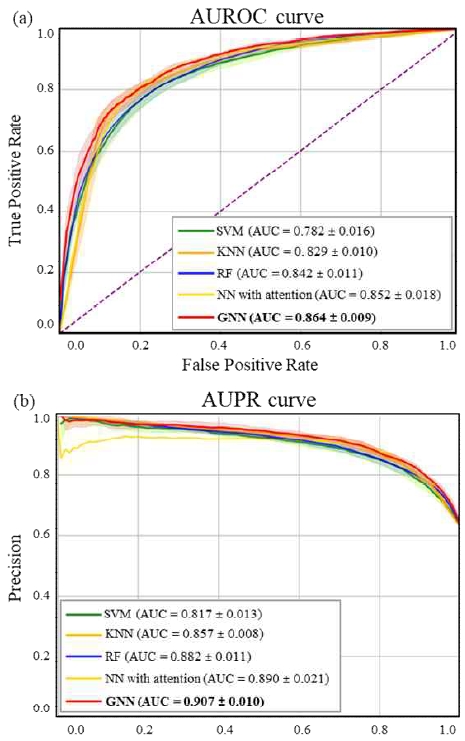
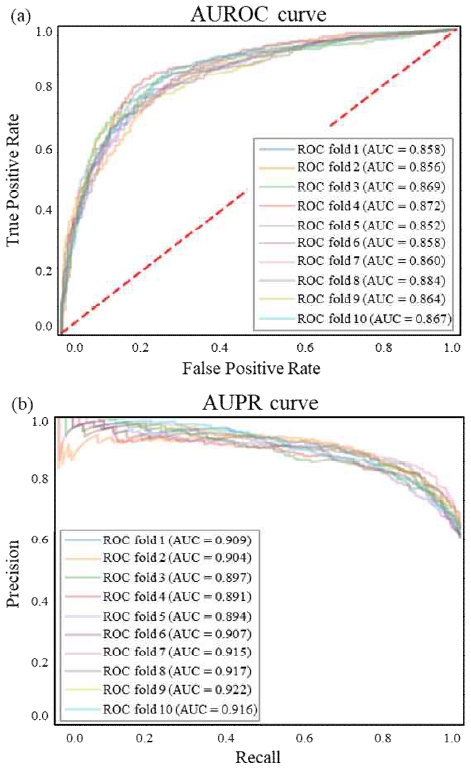
그림 4는 SVM, KNN, RF, NN with attention, GNN 모델의 AUROC와 AUPR의 성능을 보여준다. 모델의 성능을 정확하게 평가하기 위해 클래스 균형을 유지하면서 데이터를 훈련 및 테스트를 위한 하위집합으로 나누는 stratified 10-fold 교차검증을 사용하였다.
그림 4(a)는 교차검증 중 true positive rate와 false positive rate에서 도출된 ROC 곡선이다. SVM은 AUROC 성능은 0.782(±0.016), KNN은 0.829(±0.010), RF는 0.842(±0.011), NN with attention은 0.852(±0.018)을 달성하였으며 구축한 GNN 모델은 AUROC가 0.864(±0.009)을 달성하여 가장 높은 성능을 보였다. 그림 4(b)는 교차검증 중 precision과 recall의 통해 생성된 PR 곡선을 나타낸다. 여기서는 GNN이 0.907(±0.010)로 가장 높은 성능을 보였으며 그 뒤를 NN with attention이 0.890(±0.021)으로 가장 높은 성능을 보인다. SVM은 0.817(±0.013), KNN은 0.857(±0.008), RF는 0.882(±0.011)을 달성하였다.
SVM과 KNN은 각각 고정된 결계 경계를 사용하거나 지역적으로 가까운 샘플의 레이블을 참조하여 분류하는 방식이기 때문에, 화합물의 비선형적인 구조를 반영하는데 어려움이 있다. RF는 다양한 decision tree를 결합한 앙상블 학습 방법으로, 여러 개의 다양한 트리를 통해 다양한 특성을 학습할 수 있기 때문에 비선형적 데이터에서도 효과적일 수 있다. 하지만 분자의 전역적 상호작용을 포착하는 데에는 한계가 존재하기 때문에 그래프 기반 GNN보다 성능이 낮게 나타난다. NN with attention은 화합물의 중요한 특성에 집중해 예측을 수행하므로 양성 클래스의 정밀도를 높이는 데 유리하다. 하지만 동일하게 분자의 구조적 상호작용을 포착하는 데 어려움이 있기에 GNN보다 낮은 성능을 보인다. 성능을 비교하였을 때, hERG 차단제를 분류하는 데 있어, 화합물을 기반으로 그래프 구조 특성을 활용하는 것은 효과적이라고 할 수 있다. 특히, GNN은 분자의 비선형적 구조적 특성을 메시지 패싱 방식을 통해 학습할 수 있기 때문에 다른 알고리즘들에 비해 더 우수한 성능을 발휘할 수 있다[17]. 이러한 특성으로 인해 GNN은 분자 간의 복잡한 구조적 관계를 효율적으로 반영할 수 있어, hERG 차단제 예측에 있어 다른 모델들보다 더 나은 성능을 보였다.
계층화된 교차 검증은 각 폴드가 데이터 세트의 클래스 분포를 유지하여 클래스 불균형 문제를 해결할 수 있다. 이를 통해 GNN 모델은 왜곡된 데이터 분포로 인한 편항된 학습의 위험을 완화하여 모든 폴드에서 일관되게 안정적인 일반화 성능을 가진다. AUROC는 0.852에서 0.884의 범위를 보이며 AUPR 값은 0.891에서 0.922의 범위를 가진다.
3-2 그래프 신경망을 활용한 상관계수 패턴 분석
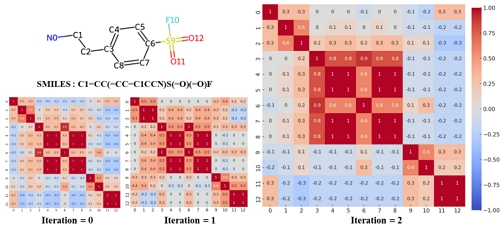
그래프 신경망을 활용하면 타겟 노드에 대해 이웃 노드와 에지의 정보를 바탕으로 업데이트한다. 훈련 중 상호작용을 분석하기 위해 각 원자 쌍의 상관계수를 히트맵으로 시각화했으며, RDkit을 활용하여 히트맵의 각 숫자는 분자 내의 원자번호를 의미한다. 벤젠 설포닐 플루오라이드의 경우, 훈련 초기(iteration = 1)와 한번 업데이트한 상태에서는 상관계수의 뚜렷한 패턴이 나타나지 않는다(그림 6). 그러나 iteration = 2에서 훈련을 진행한 후에는 히트맵에서 명확한 패턴이 나타난다.
MPNN은 거리 제약 없이 그래프 내에서 분자의 국소 정보를 전송하여 더 나은 표현이 가능하다는 장점이 있다[29]. 이를 통해 업데이트 반복 과정에서 정보가 이웃 노드에 효율적으로 전달되며, 멀리 떨어진 이웃 노드의 정보도 통합할 수 있다. 예를 들어, 원자 C3과 C5의 거리는 radius = 2이지만 훈련 전의 상관계수는 0.3이다. 이웃 노드 정보를 활용해 업데이트를 반복하면 상관계수가 0.8이 된다. 이는 그래프 신경망을 통해 실제로 상관이 있지만 거리가 멀어 낮게 평가된 상관계수를 극복할 수 있음을 보여준다.
Ⅳ. 결 론
화합물을 기반으로 한 hERG 차단제의 평가는 심장독성을 예방하는 데 중요한 역할을 한다. 이를 위해 다양한 기계학습 알고리즘과 ECFP와 같은 분자 지문이 예측에 활용되고 있다. 하지만 기계학습 방법은 주로 분자의 선형적 또는 고정된 특징 벡터에 기반한 예측을 수행하지만, 그래프 기반 모델은 노드와 에지의 비선형적 상호작용을 학습할 수 있다. 이를 위해 GNN을 활용하여 분자의 그래프 구조로부터 추출한 graphical feature를 생성하고 structural feature, physicochemical feature를 추가로 생성하여 concat한다. 이후 생성된 벡터를 입력으로 하는 FCL을 통해 hERG 채널 차단제에 대한 예측을 수행하였다.
GNN은 더 나은 예측 성능을 보이지만, 훈련 시간과 메모리 사용량 측면에서 계산 비용이 더 높다. 그러나 GNN은 SVM이나 RF와 같은 기계학습 방법으로는 얻을 수 없는 분자 그래프의 구조 정보를 캡처하므로, 예측 정확도와 심장독성 예측이라는 특성을 고려할 때, 이런 추가적인 비용은 정당화 될 수 있다. GNN 모델은 AUROC가 0.864(±0.009), AUPR이 0.907(±0.010)의 성능을 가졌으며 다른 기계학습 알고리즘과 hERG 차단제를 예측했던 NN with attention 모델보다 나은 성능을 나타낸다. 특히 그래프 기반 딥러닝 모델을 활용하여 분자의 구조적 및 화학적 특성을 효과적으로 반영하여 hERG 채널 차단제 예측에 있어 성능 향상과 신약 개발에서 심장독성에 대한 정보를 제공할 수 있어, 후보 물질을 발굴하는 도구로 활용될 수 있다.
신약 개발 초기에 화합물을 대상으로 하는 고처리 스크리닝 단계에서, 본 연구에서 제안된 GNN 모델은 hERG 차단 여부를 빠르게 예측하여 심장독성 가능성이 있는 화합물을 선별할 수 있다. 고처리 스크리닝을 통해 다수의 화합물 후보군을 선별하고, GNN 모델을 활용해 심장독성 가능성이 높은 화합물을 필터링하여 전임상 연구에 진입할 가능성이 있는 화합물을 선별하는 방식으로 통합될 수 있다. GNN 기반의 예측 모델은 기존 기계학습 모델이 캡처하지 못하는 분자의 구조적 정보와 물리화학적 특성을 고려함으로써, 더 정확한 예측과 안전한 후보 물질을 발굴하는데 중요한 도구로 활용될 수 있을 것이다.
Acknowledgments
본 연구는 전남대학교 학술연구비(과제 번호: 2021-2108)와 과학기술정보통신부 및 정보통신기획평가원의 학·석사 연계 ICT 핵심 인재 양성 사업(RS-2022-00156385), 정부(과학기술정보통신부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구(RS-2024-00332003)로서, 관계 부처에 감사드립니다.
References
-
F. De Ponti, E. Poluzzi, and N. Montanaro, “Organising Evidence on QT Prolongation and Occurrence of Torsades de Pointes with Non-Antiarrhythmic Drugs: A Call for Consensus,” European Journal of Clinical Pharmacology, Vol. 57, pp. 185-209, Jnue 2001.
[https://doi.org/10.1007/s002280100290]

-
W. S. Redfern, L. Carlsson, A. S. Davis, W. G. Lynch, I. MacKenzie, S. Palethorpe, ... and T. G. Hammond, “Relationships between Preclinical Cardiac Electrophysiology, Clinical QT Interval Prolongation and Torsade de Pointes for a Broad Range of Drugs: Evidence for a Provisional Safety Margin in Drug Development,” Cardiovascular Research, Vol. 58, No. 1, pp. 32-45, April 2003.
[https://doi.org/10.1016/S0008-6363(02)00846-5]

- J. S. Mitcheson and M. D. Perry, “Molecular Determinants of High-Affinity Drug Binding to HERG Channels,” Current Opinion in Drug Discovery & Development, Vol. 6, No. 5, pp. 667-674, November 2003.
-
B. Fermini and A. A. Fossa, “The Impact of Drug-induced QT Interval Prolongation on Drug Discovery and Development,” Nature Reviews Drug Discovery, Vol. 2, pp. 439-447, 2003.
[https://doi.org/10.1038/nrd1108]

-
C. Jamieson, E. M. Moir, Z. Rankovic, and G. Wishart, “Medicinal Chemistry of hERG Optimizations: Highlights and Hang-ups”, Journal of Medicinal Chemistry, Vol. 49, No. 17, pp. 5029-5046, June 2006.
[https://doi.org/10.1021/jm060379]

-
S. Stoelzle-Feix, A. Obergrussberger, A. Brüggemann, C. Haarmann, M. George, R. Kettenhofen, and N. Fertig, “State-of-the-Art Automated Patch Clamp Devices: Heat Activation, Action Potentials, and High Throughput in Ion Channel Screening,” Frontiers in Pharmacology, Vol. 2, 76, January 2011.
[https://doi.org/10.3389/fphar.2011.00076]

-
B. Priest, I. M. Bell, and M. Garcia, “Role of hERG Potassium Channel Assays in Drug Development,” Channels, Vol. 2, No. 2, pp. 87-93, May 2008.
[https://doi.org/10.4161/chan.2.2.6004]

-
M. K. Leong, “A Novel Approach Using Pharmacophore Ensemble/Support Vector Machine (PhE/SVM) for Prediction of hERG Liability,” Chemical Research in Toxicology, Vol. 20, No. 2, pp. 217-226, January 2007.
[https://doi.org/10.1021/tx060230c]

-
C. Cai, P. Guo, Y. Zhou, J. Zhou, Q. Wang, F. Zhang, ... and F. Cheng, “Deep Learning-based Prediction of Drug-induced Cardiotoxicity,” Journal of Chemical Information and Modeling, Vol. 59, No. 3, pp. 1073-1084, February 2019.
[https://doi.org/10.1021/acs.jcim.8b00769]

-
S. Chavan, A. Abdelaziz, J. G. Wiklander, and I. A. Nicholls, “A k-nearest Neighbor Classification of hERG K+ Channel Blockers,” Journal of Computer-Aided Molecular Design, Vol. 30, pp. 229-236, February 2016.
[https://doi.org/10.1007/s10822-016-9898-z]

-
M. R. Doddareddy, E. C. Klaasse, Shagufta, A. P. Ijzerman, and A. Bender, “Prospective Validation of a Comprehensive in Silico hERG Model and Its Applications to Commercial Compound and Drug Databases,” ChemMedChem, Vol. 5, No. 5, pp. 716-729, May 2010.
[https://doi.org/10.1002/cmdc.201000024]

-
D. Rogers and M. Hahn, “Extended-Connectivity Fingerprints,” Journal of Chemical Information and Modeling, Vol. 50, No. 5, pp. 742-754, April 2010.
[https://doi.org/10.1021/ci100050t]

-
Y. Wang, L. Huang, S. Jiang, Y. Wang, J. Zou, H. Fu, and S. Yang, “Capsule Networks Showed Excellent Performance in the Classification of hERG Blockers/Nonblockers,” Frontiers in Pharmacology, Vol. 10, 1631, January 2020.
[https://doi.org/10.3389/fphar.2019.01631]

-
A. Cavalli, E. Poluzzi, F. De Ponti, and M. Recanatini, “Toward a Pharmacophore for Drugs Inducing the Long QT Syndrome: Insights from a CoMFA Study of HERG K+ Channel Blockers,” Journal of Medicinal Chemistry, Vol. 45, No. 18, pp. 3844-3853, July 2002.
[https://doi.org/10.1021/jm0208875]

-
T. Wang, J. Sun, and Q. Zhao, “Investigating Cardiotoxicity Related with hERG Channel Blockers Using Molecular Fingerprints and Graph Attention Mechanism,” Computers in Biology and Medicine, Vol. 153, 106464, February 2023.
[https://doi.org/10.1016/j.compbiomed.2022.106464]

-
J. Y. Ryu, M. Y. Lee, J. H. Lee, B. H. Lee, and K.-S. Oh, “DeepHIT: A Deep Learning Framework for Prediction of hERG-induced Cardiotoxicity,” Bioinformatics, Vol. 36, No. 10, pp. 3049-3055, May 2020.
[https://doi.org/10.1093/bioinformatics/btaa075]

-
C. Liu, Y. Sun, R. Davis, S. T. Cardona, and P. Hu, “ABT-MPNN: An Atom-bond Transformer-based Message-passing Neural Network for Molecular Property Prediction,” Journal of Cheminformatics Vol. 15, No. 1, 29, February 2023.
[https://doi.org/10.1186/s13321-023-00698-9]

-
A. Gaulton, L. J. Bellis, A. P. Bento, J. Chambers, M. Davies, A. Hersey, ... and J. P. Overington, “ChEMBL: A Large-Scale Bioactivity Database for Drug Discovery,” Nucleic Acids Research, Vol. 40, No. D1, D1100-D1107, January 2012.
[https://doi.org/10.1093/nar/gkr777]

-
S. Kim, J. Chen, T. Cheng, A. Gindulyte, J. He, S. He, ... and E. E. Bolton, “PubChem 2023 Update,” Nucleic Acids Res, Vol. 51, No. D1, pp. D1373-D1380, January 2023.
[https://doi.org/10.1093/nar/gkac956]

-
Y. Chen, X. Yu, W. Li, and G. Liu, “In Silico Prediction of hERG Blockers Using Machine Learning and Deep Learning Approaches,” Journal of Applied Toxicology Vol. 43, No. 10, pp. 1462-1475, October 2023.
[https://doi.org/10.1002/jat.4477]

-
K.-M. Thai and G. F. Ecker, “A Binary QSAR Model for Classification of hERG Potassium Channel Blockers,” Bioorganic & Medicinal Chemistry, Vol. 16, No. 7, pp. 4107-4119, April 2008.
[https://doi.org/10.1016/j.bmc.2008.01.017]

-
D. Weininger, “SMILES, A Chemical Language and Information System. 1. Introduction to Methodology and Encoding Rules,” Journal of Chemical Information and Computer Sciences, Vol. 28, No. 1, pp. 31-36, February 1988.
[https://doi.org/10.1021/ci00057a005]

-
N. M. O’Boyle, “Towards a Universal SMILES Representation - A Standard Method to Generate Canonical SMILES Based on the InChI,” Journal of Cheminformatics, Vol. 4, 22, Septetmber 2012.
[https://doi.org/10.1186/1758-2946-4-22]

-
H. Goel, W. Yu, A. D. MacKerell, Jr., “hERG Blockade Prediction by Combining Site Identification by Ligand Competitive Saturation and Physicochemical Properties,” Chemistry, Vol. 4, No. 3, pp. 630-646, June 2012.
[https://doi.org/10.3390/chemistry4030045]

-
H. Yu, B. Zou, X. Wang, and M. Li, “Investigation of Miscellaneous hERG Inhibition in Large Diverse Compound Collection Using Automated Patch-Clamp Assay,” Acta Pharmacologica Sinica, Vol. 37, pp. 111-123, January 2016.
[https://doi.org/10.1038/aps.2015.143]

-
T. Fontanari, T. C. Fróes, and M. Recamonde-Mendoza, Cross-Validation Strategies for Balanced and Imbalanced Datasets, in Intelligent Systems, Cham: Springer International Publishing, pp. 626-640, November 2022.
[https://doi.org/10.1007/978-3-031-21686-2_43]

-
X. Zeng and T. R. Martinez, “Distribution-balanced Stratified Cross-Validation for Accuracy Estimation,” Journal of Experimental & Theoretical Artificial Intelligence, Vol. 12, No. 1, pp. 1-12, November 2010.
[https://doi.org/10.1080/095281300146272]

-
S. Prusty, S. Patnaik, and S. K. Dash, “SKCV: Stratified K-Fold Cross-Validation on ML Classifiers for Predicting Cervical Cancer,” Frontiers in Nanotechnology, Vol. 4, 972421, August 2022.
[https://doi.org/10.3389/fnano.2022.972421]

- S. Abu-El-Haija, B. Perozzi, A. Kapoor, N. Alipourfard, K. Lerman, H. Harutyunyan, ... and A. Galstyan, “MixHop: Higher-Order Graph Convolutional Architectures via Sparsified Neighborhood Mixing,” in Proceedings of the 36th International Conference on Machine Learning, pp. 21-29, April 2019. https://arxiv.org/abs/1905.00067
저자소개

2023년:전남대학교 (이학사)
※관심분야:생명정보학(bioinformatics), 인공지능(artificial intelligence)

2012년:한국한공대학교 정보통신공학과 (공학석사)
2018년:한국과학기술원 바이오및뇌공학과 (공학박사)
2018년~2019년: 국민건강보험공단 빅데이터실 부연구위원
2019년~현 재: 전남대학교 지능전자컴퓨터공학과 교수
※관심분야:생명정보학(bioinformatics), 인공지능(artificial intelligence), 빅데이터(big data) 등