엑스선 이미지 기반 결핵 예측 시스템을 위한 데이터 증강 전처리 조합법
Copyright ⓒ 2023 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
최근 헬스케어 및 의료 분야에서 컨볼루션 신경망(CNN) 모델을 활용한 X-선 이미지 분석 연구가 활발히 이루어지고 있다. X-선은 폐렴, 결핵, 유방암 등 다양한 흉부 질환을 비침습적으로 검사하는 주요 도구로서, 비용 효율적이며 많은 검사가 가능하다는 장점을 갖는다. 하지만 기존의 X-선 이미지 기반 질환 진단 인공지능 모델은 복잡한 아키텍처를 가지며 많은 파라미터와 대량의 데이터가 필요한 한계를 가진다. 본 연구에서는 이러한 문제를 해결하기 위해 적은 양의 X-선 이미지 데이터로 높은 결핵 예측 정확도를 달성할 수 있는 효과적인 전처리 조합법을 탐색하고 제안한다. 본 연구에서는 대표적인 데이터 증강법인 Random Erase, Random Flip, Random Augmentation을 함께 사용하는 전처리 조합법을 제안하였으며, 이를 ResNet50 모델과 EfficientNet-b0 모델에 적용하여 각 평균 11%, 9%의 성능 향상을 확인할 수 있었다. 또한, 전처리 조합이 적용된 모델을 쉽게 사용할 수 있도록 웹 프레임워크를 개발하였다. 사용자는 웹 프레임워크를 통해 입력 이미지를 수정할 수 있으며, 인공지능 결핵 판별 결과를 얻을 수 있다.
Abstract
Recently, there has been a surge in research within the healthcare and medical sectors that utilize Convolutional Neural Networks (CNN) to analyze X-ray images. X-rays are a primary tool for non-invasively diagnosing various thoracic diseases, including pneumonia, tuberculosis, and breast cancer. They are cost-effective and allow for extensive examinations. However, traditional AI models for disease diagnosis based on X-ray images have intricate architectures and require several parameters and vast datasets. This study addresses these issues by exploring and proposing an effective preprocessing combination method to achieve high tuberculosis prediction accuracy with limited X-ray image data. This research suggests a preprocessing combination method incorporating prominent data augmentation techniques such as Random Erase, Flip, and Augmentation. By applying this combined preprocessing to the ResNet50 and EfficientNet-b0 models, we observed an average performance improvement of 11% and 9%, respectively. Furthermore, we developed a web framework to facilitate easy access and use of the model with applied preprocessing combinations. Users can modify the input image through this web framework and obtain AI-driven tuberculosis diagnostic results.
Keywords:
Deep Learning, Health Care, Data Augmentation, X-ray Image, Web Framework키워드:
딥러닝, 헬스케어, 데이터 증강, X-선 이미지, 웹 프레임워크Ⅰ. 서 론
X-선 검사는 비용 효율적이며 비침습적인 검사 방법으로, 흉부 질환인 폐렴, 결핵, 유방암 등의 조기 발견과 치료에 필수적이다. 최근 헬스케어 및 의료 분야에서는 이러한 X-선 검사에 인공지능 활용이 증가하고 있다. 이러한 변화는 컨볼루션 신경망(CNN; Convolutional Neural Network)[1] 기반의 비전 모델 발전과 이미지 분석 연구에 높아진 관심을 배경으로 한다.
의료 도메인은 심각한 인력 부족에 시달리고있다. 이에 대응하기 위해 의사의 진단을 보조하고 조기 질병 발견이 가능한 인공지능 모델[2]-[4]의 필요성이 대두되었고, 특히 X-선 이미지를 사용한 CNN 기반 딥러닝 기법[5]-[7]을 의료 도메인에 적용하여 질환 판별, 분류, 예측하는 연구들이 활발하게 진행되고 있다.
하지만 기존의 X-선 이미지 기반 질환 진단 모델[2]-[4]은 주로 여러 비전 모델을 동시에 사용하는 방식을 채택하여 복잡한 아키텍처를 가지고있다. 여러 비전 모델의 동시 사용은 많은 훈련 데이터와 파라미터가 필요하며 데이터 수집, 라벨링, 전처리, 검증 등의 모든 과정에 많은 시간과 비용이 드는 단점을 가진다[8],[9]. 또한, 개인정보 보호와 프라이버시 이슈가 발생할 수 있는 의료 데이터의 대용량 구축은 현실적인 제약이 있을 수 있다[10].
본 논문에서는 적은 양의 X-선 이미지 데이터로 높은 결핵 예측 정확도를 달성할 수 있는 효과적인 전처리 조합법을 탐색하고 제안한다. 제안된 전처리 조합은 데이터 증강에서 주로 사용되는 Random Erase, Random Flip, Random Augmentation을 조합한것으로 ResNet50[6]과 Efficient Net-b0[7] 모델에서 정량적으로 평가되었다. 또한, 사용자가 쉽게 접근하고 사용할 수 있도록 웹 프레임워크를 개발하였다. 이 프레임워크에 우리가 제안한 인공지능 결핵 판별 모델이 포함되며, 사용자는 grad-CAM[11]정보를 제공받아 인공지능 결핵 판별 정보를 얻을 수 있다.
논문의 구성은 다음과 같다. 2장에서 이미지 처리와 데이터 전처리에 대한 기본 개념을 소개하고 의료 도메인에서 인공지능을 활용한 기존 연구들을 소개한다. 3장에서는 결핵 판별을 위해 제안한 전처리 조합법의 세부적 구성 및 실험 결과를 기술한다. 마지막 4장에서는 본 논문의 주요 결론와 향후 연구 방향에 대해 소개한다.
Ⅱ. 관련 연구 분석
2-1 CNN 기반 이미지분석
CNN[1]은 딥러닝의 한 종류로, 이미지 분석에 주로 사용되는 알고리즘이다. CNN은 이미지의 공간 정보를 보존하는 특성이 있으며 이미지에서 객체를 탐지하거나 분류하는 데 유용하다. CNN의 핵심적인 요소는 합성곱 계층(convolutional layer), 풀링 계층(pooling layer), 완전연결 계층(fully connected layer) 등이 있다. 합성곱 계층은 이미지에서 특징을 추출하며, 이를 통해 이미지 내의 패턴을 파악한다. 풀링 계층은 합성곱 계층의 출력을 다운샘플링하여 네트워크의 복잡성을 줄이고 과적합을 방지하는 역할을 한다. 완전연결 계층은 마지막 단계로 이전 계층에서 추출된 특징들을 결합하여 최종 예측을 수행한다. CNN 기반의 이미지 분석은 다양한 분야에서 활용되고 있으며, 특히 의료 이미지 분석에 효과적이다. X-선 이미지, MRI, CT 등의 이미지 데이터를 분석하여 질병을 진단하거나 예측하는 등의 작업[2]-[4]에 사용된다.
2-2 의료 도메인에서 인공지능의 활용
많은 질병 진단은 의료 영상 이미지를 바탕으로 이루어진다. 따라서, 이미지 분석에 탁월한 딥러닝 알고리즘이 질병 진단에 활용될 수 있다. 딥러닝 기반의 모델은 학습 이미지를 통해 복잡한 패턴과 특징을 학습하고, 이를 바탕으로 새로운 이미지를 분석한다. X-선 이미지 분석은 흉부 질환 진단[2]-[4], 골절 감지[12], 종양 탐지 등[13]에 널리 사용된다. 예를 들어, CNN이나 ResNet 등의 모델을 사용하여 폐렴[14], 결핵[4],[15], 유방암[16] 등의 흉부 질환을 판별하는 데 사용될 수 있다. 또한, 최근에는 X-선 이미지를 활용하여 COVID-19의 감염 여부를 판단하는 연구들도 있다[2],[3],[15].
Shah 등[17]은 X-선 및 CT 이미지를 사용하여 COVID-19를 감지하는 데 인공지능 기반 시스템을 사용하였다. 이 논문에서는 총 80개의 논문을 검토하고 데이터 세트, 전처리 기법, 분할 방법, 특징 추출, 분류, 실험 결과를 탐색하고 분석하여 인공지능 기반 프레임워크를 제안하였다. Khan 등[18]은 COVID-19 진단을 위해 전이학습으로 훈련시킨 4개의 딥러닝 모델[5],[6],[19]을 비교하였다. Ghaderzadeh 등[15]은 COVID-19 진단을 위해 개발된 딥러닝 모델을 서베이 논문으로 정리하였다.
Rahman 등[4]은 U-Net[20] 기반 모델을 사용하여 폐 X-선 이미지를 세그먼트하고 결핵에 걸린 폐를 분류하는 모델을 제안하였다. Yadav 등[21]은 Coarse-to-Fine 접근법을 사용하여 이미지 데이터 부족 문제를 해결하고 GoogleNet[22] 기반 모델을 사용하여 잠재적 결핵 환자를 분류하는 프레임워크를 제안하였다.
딥러닝 모델은 전문가 수준의 성능을 보여주며, 일부는 전문가를 능가하는 예측 결과를 보여준다. 하지만, 기존 딥러닝 기반 X-선 이미지 분석 모델은 많은 훈련 데이터와 높은 계산 능력을 필요하며 모델 최적화에 오랜 시간이 소요되는 문제점이 있다. 또한, 의료 데이터의 개인 정보보호 문제로 공개 데이터셋을 찾기 어려우며 데이터셋의 크기 또한 충분치 않은 경우가 많다. 해결 방법으로 전이 학습(Transfer Learning)[23]과 데이터 증강을 통한 모델 훈련이 있으며 본 논문에서는 데이터 증강을 통해 문제를 해결한다.
2-3 데이터 증강
데이터 증간은 전처리 방법중 하나이다. 이미지 데이터 증강은 적은 데이터 양으로 높은 성능을 얻기 위한 방법이다. 기존의 이미지 데이터를 변형하거나 확장하여 데이터셋의 다양성을 증가시키고 모델 성능 향상, 과적합 방지등의 장점을 갖게한다.
Shorten 등[24]은 다양한 이미지 데이터의 증강법을 조사하였다. 최근 가장 많이 사용되는 이미지 데이터 증강법은 기하학적 변환법(Random Flip, Random Crop 등), 색 공간 증강, 커널 필터, 이미지 혼합, 무작위 지우기(Random Erase)[25], 특징 공간 증강 등으로 조사되었다.
기하학적 변환 방법론 중 하나인 Random Flip은 이미지를 일정 확률로 수평 또는 수직으로 뒤집는다. 이 방법은 Simonyan 등[5]에 의해 모델 훈련에 활용되었다.
Random Crop은 이미지를 무작위 크기로 잘라내는 방식으로 데이터를 증강한다. 이 방법은 Alex 등[26]이 제안한 모델을 훈련시키기 위한 데이터 증강에 활용되었다. 또한 Taylor 등[27]의 연구는 간단한 CNN 네트워크에서 Random Crop을 적용하였을 경우에 좋은 성능 향상을 기대할 수 있다는 것을 보여주었다.
Random Erase는 이미지의 직사각형 영역을 무작위로 선택하여 해당 영역을 지우거나 노이즈를 추가하는 방식으로 데이터를 증강한다. Zhong 등[25]에 의해 제안된 이 방법을 다양한 CNN 아키텍처[5],[6]에 적용하여 모델의 성능 향상이 확인할 수 있었다.
앞서 소개된 다양한 데이터 증강 방법론을 조합하여 사용하는 Random Augmentation 기법도 연구되었다[28]-[30]. 그러나 증강 방법론의 수가 늘어날수록 적합한 증강 파라미터를 찾는 데 필요한 시간과 비용이 증가하는 문제가 발생한다. 이 문제를 해결하기 위해 Cubuk 등[31]은 최적화된 검색 공간을 탐색하는 그리드 알고리즘을 제안하였다.
이렇게 증강된 이미지 데이터는 실세계의 다양한 시나리오를 반영할 수 있어 데이터셋의 다양성을 향상시키는 것이다. 예를 들어, 실제 세상에서는 같은 사물이나 사람이 다양한 각도에서 보이거나, 다양한 조명 하에서 보일 수 있다. 데이터 증강은 이러한 실세계의 다양성을 고려하여 데이터 변형으로 증강하고 모델이 학습할 수 있도록 한다. 이를 통해, 모델은 훈련 데이터에 과적합되는 것을 막고, 실세계의 다양한 상황에서도 잘 동작할 수 있다.
Ⅲ. 설계 및 실험결과
3-1 데이터셋 분석
본 연구에서 Tuberculosis Chest X-ray Database[4]와 Tuberculosis Chest X-rays(Shenzhen)[32] 데이터베이스를 사용하여 데이터셋을 구축하였다. 데이터셋은 8:2의 비율로 훈련 데이터와 테스트 데이터로 분할하여, 총 4900장의 이미지 중 결핵 훈련 이미지는 3920장, 테스트 이미지는 980장이 포함되었다. 이러한 비율 설정은 충분한 모델 학습과 객관적 평가가 가능한 테스트 데이터 확보를 위함이다[33],[34]. 표 1은 데이터셋의 분포를, 그림 1은 데이터의 상태를 보여준다. 모델에서 처리되는 이미지 크기를 고려하여 데이터셋의 이미지는 512×512 픽셀로 스케일링하였다. 원래 데이터셋 이미지는 32bit RGB형식이었으나, 모델에서는 OpenCV 라이브러리를 활용하여 255 grayscale로 변환하였다. 이 변환 과정은 폐의 음영과 조직을 더욱 선명하게 보여주어 이미지 분석에 정확성을 높일 수 있다.
3-2 아키텍쳐
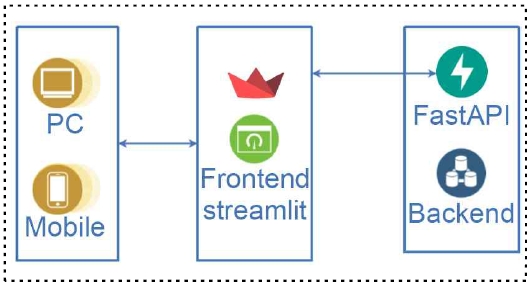
전체적인 시스템의 아키텍처는 그림 2에 표현되어 있다. 본 시스템은 프론트엔드와 백엔드로 나뉘어져 있으며 프론트엔드는 Streamlit을 사용하여 환자의 X-선 사진을 PC 또는 모바일 디바이스에서 입력 받을 수 있는 구조로 구현되었다.
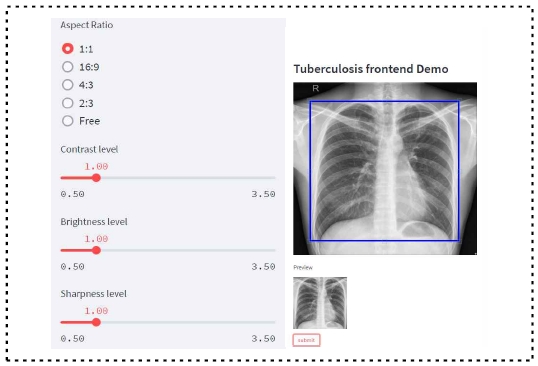
시스템을 시작하면 사용자에게 그림 3과 같은 인터페이스가 제공된다. 사용자는 사이드바 및 크롭박스를 사용하여 X-선 이미지의 크기, 명도, 밝기, 선명도 등을 변경하여 입력할 X-선 이미지를 원하는 대로 수정하고 입력한다.
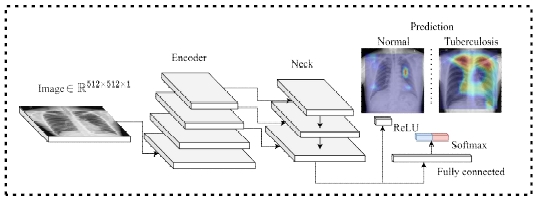
X-선 이미지 입력 후, 시스템은 백엔드에서 PyTorch 기반 X-선 이미지 분류 모델을 실행한다. 그림 4는 이에 대한 모델 스키마를 보여준다. 입력된 이미지는 512×512×1 크기의 Grayscale 이미지로 변환되고 이미지 인코더에 전달되어 저차원 공간에 임베딩된다. 이미지 인코더는 FPN(feature-pyramid network)[35] 기반 ‘Neck’ 구조를 활용하여 이미지에 대한 피쳐를 보강한다. 이 보강된 피쳐들은 Fully Connected Layer와 softmax 함수를 거쳐서 결핵 여부를 예측한다. 예측 후 사용자 인터페이스에는 결핵 여부, confidence score, 그리고 Grad-CAM을 통해 분석된 결과사진을 확인할 수 있다. 이 결과 사진에 제공된 시각적 정보는 모델이 입력 이미지의 어느 부분에 주목하여 결핵 판별을 하였는지 보여준다. 이러한 정보 제공은 블랙박스로 느껴지는 모델의 작동 방식을 사용자가 이해하는데 도움을 줄 수 있다.
3-3 전처리 조합에 따른 결과 분석
본 연구에서는 ResNet50[6]과 EfficientNet-b0[7]에 데이터 증강 전처리 과정을 추가하였다. 이 모델들은 이미지를 기반으로 학습하는 모델이며 서로 다른 모델 구조를 가진다. 본 연구에서 적용한 전처리 과정은 그림 5-7에 의사코드로 제공되어 있으며, 전처리 과정은 각 훈련 데이터 이미지에 적용되어 데이터 증강에 사용되었다. 전처리된 이미지들은 ResNet50과 EfficientNet-b0 모델에 각각 입력되어 학습이 진행되었다. 두 모델은 동일한 학습 데이터를 사용하였으며 전처리의 성능을 평가하기 위해 독립적으로 학습되었다. 모델의 성능 평가는 정확도(Accuracy), 정밀도(Precision), 재현율(Recall), F1-Score의 4가지 척도를 사용하여 진행되었으며 Accuracy는 모델이 올바른 예측을 한 비율을 나타내는 수식으로 다음 식 (1)과 같다.
| (1) |
Precision은 모델이 True라고 예측한 경우 중 실제로 True인 비율을 나타내며 수식으로는 다음 식 (2)와 같다.
| (2) |
Recall은 실제 True인 케이스 중 모델이 True라고 예측한 비율을 나타내며 수식으로는 다음 식 (3)과 같다.
| (3) |
F1-Score는 Precision과 Recall의 조화 평균을 나타내고 수식으로는 다음 식 (4)와 같다.
| (4) |
데이터 증강 전처리 과정을 통해 얻은 데이터로 훈련된 모델 성능 평가는 표 2와 같다. ResNet50 모델의 경우, 전처리를 적용하지 않았을 때의 정확도는 85.6%, 정밀도는 78.2%, 재현율은 90.0%, F1-Score는 81.1%였다. 하지만, 'RandErasing' [25], 'RandAugment'[31], 'RandFlip'[5] 전처리를 순차적으로 적용하면 모든 성능 지표가 개선되었다. 특히, 세 가지 전처리 모두를 적용했을 때의 정확도는 96.3%, 정밀도는 93.6%, 재현율은 94.6%, F1-Score는 94.1%으로 개선되어 가장 높은 성능을 보였다. EfficientNet-b0 모델 역시 전처리를 적용하였을 때 정확도가 85.9%에서 94.2%, 정밀도는 78.0%에서 89.8%, 재현율은 88.0%에서 92.3%, F1-Score는 80.9%에서 91.0%으로 각각 증가하는 것을 확인할 수 있었다.
본 연구의 결과는 ResNet50과 EfficientNet-b0 모델을 사용하여 X-선 이미지 분석을 진행하였을 경우 제안한 전처리 조합이 모델의 성능을 개선하는 것을 확인하였다. 특히 Random Erase나 Random Flip같은 기하학적인 증강과 Random Augment 같은 광학적인 증강을 같이 사용하였을 때 가장 좋은 성능 향상을 보여주었다.
3-4 세부 구현
본 연구에서는 싱글 NVIDIA GTX 3090 GPU를 사용하여모델 학습을 진행하였다. 학습 데이터셋에 대해 총 10 에포크를 진행하였으며, 한 번에 32개의 이미지를 포함하는 배치 크기를 사용하였다. 모델의 파라미터 업데이트를 위해 AdamW[36] 옵티마이저를 사용하였다. AdamW는 Adam 알고리즘에 가중치 감소를 도입한 것으로, 모델의 일반화 능력을 향상시키는 데 도움이 된다. 옵티마이저의 학습률(lr)은 0.001로 설정하였으며, 가중치 감소율(weight decay)은 0.05로 설정하였다. 모델에 입력되는 모든 X-선 이미지는 정규화 과정을 거쳤으며, 평균(mean)은 [123.675, 116.28, 103.53], 표준편차(std)는 [58.395, 57.12, 57.375]로 설정하였다. 정규화 이후, 모든 이미지는 opencv라이브러리의 cvtColor 함수를 사용하여 그레이스케일로 변환한다. RandomErasing의 setting_prob는 0.5, area_ratio는 0.02~0.4, aspect_range는 0.3~3.3 으로 설정하였다. Random Augment의 num_policies는 2, Random Flip의 setting_prob는 0.5으로 설정하였다.
Ⅳ. 결론 및 향후 연구
본 연구에서는 제한된 양의 X-선 이미지 데이터셋을 사용하여도 높은 결핵 예측 정확도를 달성할 수 있는 효과적인 전처리 조합법을 제안하였다. 실험 결과, ResNet50 모델은 전처리를 적용할 경우 평균 11%의 성능 향상되었고 EfficientNet-b0 모델은 전처리 후 평균 9%의 성능 향상이 있음을 확인하였다. 이 결과는 우리가 제안한 전처리 조합법이 X-선 결핵 이미지를 분석하는 모델의 성능을 향상시키는 데 효과적임을 입증한다.
또한, 제시한 전처리 조합법이 적용된 이미지 처리 모델을 쉽게 사용할 수 있도록 사용자 인터페이스를 개발하였다. 향후 연구에서는 제시한 전처리 조합법을 다양한 의료 이미지 데이터셋이나 다른 도메인의 이미지 데이터셋에 확대 적용하여 성능 향상에 도움이 되는지 알아보겠다. 또한, 본 연구에서 제안한 전처리 조합법의 효과를 최대화할 수 있는 다른 모델 구조나 학습 전략에 대한 연구도 필요하다.
Acknowledgments
이 논문은 전남대학교 학술연구비(과제번호: 2023-0483-01) 지원과 과학기술정보통신부 및 정보통신기획평가원의 지역지능화혁신인재양성사업의 연구결과로 수행되었음(IITP-2023-RS-2022-00156287)
References
-
P. Y. Simard, D. Steinkraus, and J. C. Platt, “Best Practices for Convolutional Neural Networks Applied to Visual Document Analysis,” in Proceeding of Seventh International Conference on Document Analysis and Recognition, Edinburgh, UK, pp. 958-963, August 2003.
[https://doi.org/10.1109/icdar.2003.1227801]

-
L. Kong and J. Cheng, “Classification and Detection of COVID-19 X-Ray Images Based on DenseNet and VGG16 Feature Fusion,” Biomedical Signal Processing and Control, Vol. 77, 103772, August 2022.
[https://doi.org/10.1016/j.bspc.2022.103772]

-
A. Shelke, M. Inamdar, V. Shah, A. Tiwari, A. Hussain, T. Chafekar, and N. Mehendale, “Chest X-ray Classification Using Deep Learning for Automated COVID-19 Screening,” SN Computer Science, Vol. 2, No. 4, 300, 2021.
[https://doi.org/10.1007/s42979-021-00695-5]

-
T. Rahman et al., “Reliable Tuberculosis Detection Using Chest X-Ray With Deep Learning, Segmentation and Visualization,” IEEE Access, Vol. 8, pp. 191586-191601, 2020.
[https://doi.org/10.1109/access.2020.3031384]

-
K. Simonyan and A. Zisserman, “Very Deep Convolutional Networks for Large-Scale Image Recognition,” arXiv, 2014,
[https://doi.org/10.48550/arxiv.1409.1556]

-
K. He, X. Zhang, S. Ren, and J. Sun, “Deep Residual Learning for Image Recognition,” in Proceeding of 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), pp. 770-778, 2016.
[https://doi.org/10.1109/cvpr.2016.90]

-
M. Tan and Q. V. Le, “EfficientNet: Rethinking Model Scaling for Convolutional Neural Networks,” arXiv, 2019,
[https://doi.org/10.48550/arxiv.1905.11946]

- L. Alzubaidi, J. Zhang, A. J. Humaidi, A. Al-Dujaili, Y. Duan, O. Al-Shamma, ... and L. Farhan, “Review of Deep Learning: Concepts, CNN Architectures, Challenges, Applications, Future Directions,” Journal of Big Data, Vol. 8, 53, pp. 1-74.
-
I. H. Sarker, “Deep Learning: A Comprehensive Overview on Techniques, Taxonomy, Applications and Research Directions,” SN Computer Science, Vol. 2, No. 6, 420, 2021.
[https://doi.org/10.1007/s42979-021-00815-1]

-
W. N. Price and I. G. Cohen, “Privacy in the Age of Medical Big Data,” Nature Medicine, Vol. 25, No. 1, pp. 37-43, 2019.
[https://doi.org/10.1038/s41591-018-0272-7]

- R. R. Selvaraju, M. Cogswell, A. Das, R. Vedantam, D. Parikh, and D. Batra, “Grad-Cam: Visual Explanations from Deep Networks via Gradient-Based Localization,” in Proceeding of the IEEE International Conference on Computer Vision(ICCV), pp. 618-626, 2017.
- L. Tanzi, E. Vezzetti, R. Moreno, and S. Moos, “X-ray Bone Fracture Classification Using Deep Learning: A Baseline for Designing a Reliable Approach,” Vol. 10, No. 4, p. 1507.
-
W. Ausawalaithong, A. Thirach, S. Marukatat, and T. Wilaiprasitporn, “Automatic Lung Cancer Prediction from Chest X-ray Images Using the Deep Learning Approach,” in Proceedings of 11th Biomedical Engineering International Conference (BMEiCON), Chiang Mai, Thailand, 2018.
[https://doi.org/10.1109/BMEiCON.2018.8609997]

- E. Ayan and H. M. Ünver, “Diagnosis of Pneumonia from Chest X-ray Images Using Deep Learning,” in Proceeding of 2019 Scientific Meeting on Electrical-Electronics & Biomedical Engineering and Computer Science (EBBT), Istanbul, Turkey, 2019.
-
M. Ghaderzadeh and F. Asadi, “Deep Learning in the Detection and Diagnosis of COVID-19 Using Radiology Modalities: A Systematic Review,” Journal of Healthcare Engineering, Vol. 2021, 6677314, 2021.
[https://doi.org/10.1155/2021/6677314]

- E. Rashed and M. S. A. El Seoud, “Deep Learning Approach for Breast Cancer Diagnosis,” in Proceedings of the 8th International Conference on Software and Information Engineering, pp. 243-247, April 2019.
-
F. M. Shah, S. K. S. Joy, F. Ahmed, T. Hossain, M. Humaira, A. S. Ami, ... and S. Ahmed, “A Comprehensive Survey of COVID-19 Detection Using Medical Images,” SN Computer Science, Vol. 2, No. 6, 434, 2021.
[https://doi.org/10.1007/s42979-021-00823-1]

-
I. U. Khan and N. Aslam, “A Deep-Learning-Based Framework for Automated Diagnosis of COVID-19 Using X-ray Images,” Information, Vol. 11, No. 9, 419, 2020.
[https://doi.org/10.3390/info11090419]

-
G. Huang, Z. Liu, L. V. D. Maaten, and K. Q. Weinberger, “Densely Connected Convolutional Networks,” in Proceedings of 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), pp. 2261-2269, 2017.
[https://doi.org/10.1109/cvpr.2017.243]

-
O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,” in Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention, 2015.
[https://doi.org/10.48550/arXiv.1505.04597]

-
O. Yadav, K. Passi, and C. K. Jain, “Using Deep Learning to Classify X-ray Images of Potential Tuberculosis Patients,” in Proceedings of 2018 IEEE International Conference on Bioinformatics and Biomedicine (BIBM), Madrid, Spain, pp. 2368-2375, 2018.
[https://doi.org/10.1109/BIBM.2018.8621525]

-
C. Szegedy, W. Liu, Y. Jia, P. Sermanet, S. Reed, D. Anguelov, ... and A, Rabinovich, “Going Deeper with Convolutions,” in Proceeding of 2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Boston, MA, 2015.
[https://doi.org/10.1109/cvpr.2015.7298594]

-
K. He, R. Girshick, and P. Dollár, “Rethinking ImageNet Pre-training,” arXiv, 2018.
[https://doi.org/10.48550/arxiv.1811.08883]

-
C. Shorten and T. M. Khoshgoftaar, “A Survey on Image Data Augmentation for Deep Learning,” Journal of Big Data, Vol. 6, 60, 2019.
[https://doi.org/10.1186/s40537-019-0197-0]

-
Z. Zhong, L. Zheng, G. Kang, S. Li, and Y. Yang, “Random Erasing Data Augmentation,” arXiv, 2017,
[https://doi.org/10.48550/arxiv.1708.04896]

-
A. Krizhevsky, I. Sutskever, and G. E. Hinton, “ImageNet Classification with Deep Convolutional Neural Networks,” Communications of the ACM, Vol. 60, No. 6, pp. 84-90, 2017.
[https://doi.org/10.1145/3065386]

-
L. Taylor and G. Nitschke, “Improving Deep Learning with Generic Data Augmentation,” 2018 IEEE Symposium Series on Computational Intelligence (SSCI), Bangalore, India, 2018.
[https://doi.org/10.1109/ssci.2018.8628742]

-
E. D. Cubuk, B. Zoph, D. Mane, V. Vasudevan, and Q. V. Le, “AutoAugment: Learning Augmentation Policies from Data,” arXiv, 2018.
[https://doi.org/10.48550/arxiv.1805.09501]

-
D. Ho, E. Liang, I. Stoica, P. Abbeel, and X. Chen, “Population Based Augmentation: Efficient Learning of Augmentation Policy Schedules,” arXiv, 2019.
[https://doi.org/10.48550/arxiv.1905.05393]

-
S. Lim, I. Kim, T. Kim, C. Kim, and S. Kim, “Fast AutoAugment,” arXiv, 2019,
[https://doi.org/10.48550/arxiv.1905.00397]

-
E. D. Cubuk, B. Zoph, J. Shlens, and Q. V. Le, “RandAugment: Practical Automated Data Augmentation with a Reduced Search Space,” arXiv, 2019.
[https://doi.org/10.48550/arxiv.1909.13719]

- S. Jaeger, S. Candemir, S. Antani, Y.-X. J. Wáng, P.-X. Lu, and G. Thoma, “Two Public Chest X-ray Datasets for Computer-aided Screening of Pulmonary Diseases,” Vol. 4, No. 6, p. 475.
- V. R. Joseph, “Optimal Ratio for Data Splitting,” Vol. 15, No. 4, pp. 531-538.
- P. Baheti, “Train Test Validation Split: How To & Best Practices [2022]”.
-
A. Kirillov, R. Girshick, K. He, and P. Dollar, “Panoptic Feature Pyramid Networks,” 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR), Long Beach, CA, June 2019.
[https://doi.org/10.1109/cvpr.2019.00656]

-
I. Loshchilov and F. Hutter, “Decoupled Weight Decay Regularization,” arXiv, 2017.
[https://doi.org/10.48550/arxiv.1711.05101]

저자소개

2021년:전남대학교 학사
2022년:전남대학교 대학원(공학석사 - 인공지능융합)
2022년~현 재: 전남대학교 인공지능융합학과 석사과정
※관심분야:딥러닝, 증강/가상현실, 인간-컴퓨터 상호작용

2009년:University of Tasmania 학사
2011년:University of Tasmania 석사
2016년:University of Canterbury 박사
2016년~2020년: University of South Australia 박사 후 연구원
2017년~2020년: Swinburne University of Technology 박사 후 연구원
2020년~2021년: 한국과학기술연구원 박사 후 연구원
2021년~현 재: 전남대학교 인공지능학부 조교수
※관심분야:증강/가상현실, 인간-컴퓨터 상호작용, 딥러닝