웹 기반 단일염기다형성 연관 패스웨이 분석 도구
Copyright ⓒ 2018 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
질환의 원인을 규명하기 위해 전장유전체 연관분석 (GWAS; Genome-Wide Association Study) 연구가 활발히 진행되고 유전체 레벨의 단일염기다형성 (SNP; Single-nucleotide polymorphism)이 많이 밝혀지고 있다. 그러나 단일염기다형성의 연관분석을 통해 질환이 발병하는 생물학적 메카니즘을 이해하기 어렵기 때문에 유전자, 생물학적 패스웨이 및 질환 등의 연관성 분석이 이전보다 더욱 중요하다. 본 논문에서는 단일염기다형성과 관련된 유전자와 패스웨이, 질환 정보를 검색하여 통합 분석하는 서비스를 제공하는 PRaDA 웹 시스템을 제안하였다. PRaDA는 사용자로부터 입력받은 유의한 몇몇의 단일염기다형성들과 관련된 유전자 및 패스웨이 뿐만 아니라, 유의하지 않은 다수의 단일염기다형성 집합의 간접적인 영향을 파악하기 위해 기능적으로 근접한 패스웨이를 검색하고 통계적 분석을 실행한다. 사용자들은 PRaDA가 제공하는 통합된 정보를 통해 질병의 전반적인 이해를 할 수 있다.
Abstract
Genome-Wide Association Study (GWAS) have been used to identify susceptibility genes for complex human diseases and many recent studies succeed to report common genetic factors for various diseases. Unfortunately, it is hard to understand all biological functions and mechanisms around the complex disease with GWAS only although the number of known associated genes with diseases is increased drastically because GWAS is a single locus based approach while not a gene but numerous factors may affect a disease associated pathways. PRaDA generates a combined report with genes, pathways and Gene Ontology (GO) using single nucleotide polymorphism (SNP) analysis output. The PRaDA reports not only directly associated pathways but also functionally related ones for identifying accumulated effects of low p-value SNPs. Through integrated information including indirect functional effects, user could have insights of overall disease mechanisms and markers.
Keywords:
SNP, GWAS, Gene interaction, Biological pathway, Gene set enrichment analysis키워드:
단일염기다형성, 전장유전체 연관분석, 유전자 상호작용, 유전자 집합 분석, 생물학적 패스웨이, 복합 질환Ⅰ. 서론
근래의 전장유전체 연관분석 (GWAS) 연구는 각 질환의 환자들에서 발견되는 공통된 유전적 변이와의 관계를 밝히기 위해 더욱 활발하게 진행되고 있으나, 여전히 그 결과로부터 생물학적 의미를 유출하고 관련된 생물학적 패스웨이와 질환의 복합적 현상을 이해하는 것은 어려운 문제이다[1]-[9]. 따라서 유전체 레벨의 단일염기다형성(SNP) 분석을 위한 유전자와 패스웨이, 질환의 연관성 분석과 그 방법론의 필요성이 대두되고 있다. 일반적인 접근방법은 가장 유의한 몇몇 단일염기다형성들을 분석하지만, 실제 유의한 단일염기다형성은 복합 질환에 미약한 영향을 끼치거나 질환과의 연관성을 밝히는 데 한계가 있다[4]-[6]. 유의성이 낮은 단일염기다형성은 여러 개의 다른 단일염기다형성들 또는 유전자들과 함께 작용하여 질환을 일으키는데 중요한 역할을 하기도 한다[8]. 또한 단일염기다형성이 질환에 직접적인 영향을 미치지는 않지만, 여러 패스웨이를 거쳐 질환과 유의하게 연관된 유전자 발현에 영향을 미침으로써 질환 발병에 관여하는 경우도 보고되었다[10]-[13]. 따라서 단일염기다형성과 유전자, 패스웨이, 질환 정보 등의 복합적인 연관 분석이 중요하다.
단일염기다형성과 생물학적 패스웨이 간의 연관 분석 방법을 제시한 서비스로 i-Gseas4Gwas[9], ICSNPPathway[14], DAVID[11], SNPtoGO[15] 등이 있다. 그러나 하나의 단일염기다형성을 이용하여 분석하거나, 다수의 단일염기다형성이 위치하는 유전자와 관련된 다수의 패스웨이와 질환간의 통합 분석이 용의하지 않다 (Table 1). 대부분의 프로그램들은 단일염기다형성을 이용한 키워드 검색 기능과 관련 정보를 각각 분리하여 제공한다. 또한 검색 결과 다운로드 기능의 부재로 검색 결과를 분석하기 위한 다음 단계에 사용할 수 없으며, 일시적인 정보로 효용 가치가 떨어지고 부가적인 데이터 수집 및 취합의 작업이 필요하다.
본 논문에서는 다수의 단일염기다형성과 관련된 유전자와 패스웨이, 질환 정보를 통합적으로 검색하고 분석할 수 있는 서비스인 PRaDA를 제안하였다. PRaDA는 사용자로부터 입력받은 dbSNP (Single Nucleotide Polymorphism Database) [16] 기반 아이디를 기준으로 유전자와 패스웨이, 질환 정보를 제공하고, 유전자 집합 분석(GSEA; Gene Set Enrichment Analysis) 뿐만 아니라 단일염기다형성 집합 분석 (SSEA; SNP Set Enrichment Analysis)을 실행하여 질환의 유의한 생물학적 지표를 밝히는데 필요한 정보를 제공한다. 기존의 분석 도구[9],[11],[14],[15]들보다 더 많은 패스웨이 정보를 포함하는 PRaDA의 데이터베이스를 구축하고 이를 기반으로 분석이 이루어지기 때문에 PRaDA는 더 다양하고 종합적인 결과를 산출할 수 있다.
Ⅱ. 본 론
2-1 시스템 환경
PRaDA는 오픈소스 소프트웨어를 기반으로 구현된 시스템이다. JSP/Servlet 기술이 사용되었으므로 SUN Microsystem에서 제공하는 버전 1.6 이상의 Java 가상머신 혹은 그에 준하는 호환 컴파일러가 필요하다. 또한 Servlet으로 구현된 웹서비스를 위한 어플리케이션 서버에는 Apache의 Tomcate을 사용하였고, 데이터베이스 엔진에는 공개프로그램인 MySQL을 사용하였다. PRaDA의 구동에는 별도의 어플리케이션 혹은 데이터베이스 서버를 두는 분산형 클라이언트-서버 환경을 권장하지만, 불가피한 경우에는 표준 사이즈의 데스크탑 PC 단독만으로도 서비스를 실행할 수 있다.
2-2 데이터
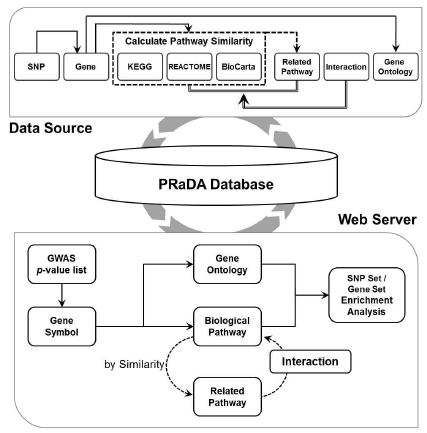
단일염기다형성이 위치하는 유전자 정보를 기반으로 관련된 패스웨이와 Gene Ontology (GO) 등의 정보를 검색하기 위해 공개 데이터를 바탕으로 PRaDA 데이터베이스를 구축하였다(Fig. 1). dbSNP와 refGene[17]은 최신 버전보다 지금 상황에 있어서 연구에 많이 사용되는 dbSNP 131과 refGene human geneom 19 버전을 선택하였고, 패스웨이와 GO는 최신 버전을 사용하였다. dbSNP 131은 26,033,053개 SNP IDs 정보를 제공하고 refGene hg19은 22,157개 종류의 유전자 심볼(symbol)에 해당하는 37,231 유전자 아이디를 포함한다. 그러나 단일 또는 다중 염색체에서 여러 개의 위치 정보를 가지거나, 정확하게 염색체 정보가 할당되지 않은 단일염기다형성과 유전자는 분석에서 제외되었다. 생물학적 패스웨이 데이터는 BioCarta와 REACTOME[18]을 재정리한 MSigDB[19]와 KEGG[20] 데이터베이스를 사용하였다. PRaDA 데이터베이스는 KEGG의 244개 휴먼 패스웨이, 그와 관련된 5,795 종류의 유전자 심볼 정보, 기능적으로 관련된 패스웨이 정보를 포함한다. 그리고 MSigDB는 BioCarta와 REACTOME 데이터베이스만큼 잘 정리된 KEGG 정보도 제공하지만, 실제 KEGG의 데이터보다 적은 5,222개 유전자와 관련된 186개 패스웨이만을 포함한다. 또한 KEGG에서만 제공하는 패스웨이와 생물학적 기능적으로 관련된 패스웨이 (Related Pathway) 정보는 MSigDB에 정리되어 있지 않기 때문에, 우리가 직접 KEGG 정보를 추출하여 정리하여 제공한다. GO 데이터는 34,250개 용어(term)의 특징과, 3가지 생물학적 기능 관점 (cellular components, biological processes, and molecular functions)에 따른 용어 타입 정보를 추출하였다. PRaDA는 각 생물학적 패스웨이와 GO에 관련된 유전자와 단일염기다형성의 개수를 사전에 계산한 테이블을 생성하여 유전자 집합과 단일염기다형성 집합의 통계적 분석 (GSEA; Gene-set Enrichment Analysis, SSEA; SNP-set Enrichment Analysis)을 실행한다.
2-3 모듈
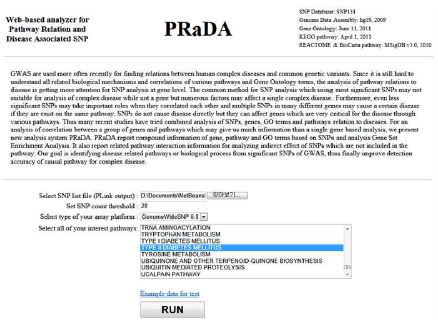
PRaDA는 GWAS 결과를 이용하여 단일염기다형성의 생물학적 기능을 밝히기 위해, 클라이언트로부터 GWAS 결과 파일과 단일염기다형성의 p-value 임계치 (threshold), 관심있는 패스웨이 리스트, 단일염기다형성 어레이 플랫폼 (SNP array platform) 정보를 입력 받는 인터페이스(Fig. 2)와 7개의 모듈로 구성된 시스템이다. p-value 임계치를 기준으로 입력받은 단일염기다형성을 필터링하여 유의한 단일염기다형성을 포함하는 유전자 정보뿐만 아니라 유전자가 참여하는 패스웨이, 유전자의 생물학적 기능 (molecular functions, biological processes, cellular components) 등의 정보를 검색한다. 또한 클라이언트의 관심 패스웨이 리스트와 실제 단일염기다형성이 위치하는 패스웨이 리스트의 비교 분석이 가능하고, 각 패스웨이와 GO 용어를 기준으로 단일염기다형성 집합의 통계적 분석 (Enrichment analysis)를 실행한다. 이 모듈들은 단계적, 병렬적, 또는 선택적으로 실행된다.
웹 기반 인터페이스에서 입력받은 단일염기다형성 아이디와 p-value로 구성된 전장유전체 연관분석 결과 파일과 p-value 임계치를 통해 무의미한 단일염기다형성을 제외하여 적정한 수준의 단일염기다형성을 선택적으로 필터링하는 모듈이다. 단일염기다형성이 위치하는 유전자를 검색하고, 다음 모듈에 사용할 수 있도록 가장 유의한 단일염기다형성부터 정렬하여 아이디 목록을 생성한다.
이전 단계에서 생성한 여러 단일염기다형성들이 유전체상에서 위치하는 유전자를 검색한다. 단일염기다형성이 존재하는 염색체와 위치 정보를 dbSNP 테이블로부터 추출하고, 유전자의 염색체와 전사 영역 (transcription region)을 refGene 테이블로부터 추출하여 매핑한다. refGene 테이블에는 ID 기준으로 유전자에 대한 정보가 저장되어 있지만, 패스웨이와 GO 관련 데이터베이스와의 매핑을 유전자 심볼을 기준으로 이루어지기 때문에 유전자 심볼 정보를 추출하여 유전자 리스트를 생성한다.
PRaDA의 내부 데이터베이스는 패스웨이 정보 검색에 필요한 두 개의 테이블을 가진다. 첫 번째 패스웨이 테이블은 KEGG의 패스웨이 아이디와 패스웨이 이름, 특징, GO 등의 정보를 담고 있다. 두 번째 패스웨이-유전자 테이블은 각 패스웨이에 참여하고 있는 유전자와 유전자 특징 정보를 제공한다. 수십 개 이상의 유전자가 기능적으로 참여하고 있는 패스웨이 127,456개가 있기 때문에, 사용자 목적에 따른 데이터 양과 빠른 비교 검색을 위해 KEGG 테이블을 분리하여 저장하였다. 인간(human) 외의 유기체(organism)에서 밝혀진 패스웨이 정보가 테이블에 포함되어 있기 때문에, 불필요한 검색작업을 피하기 위해 다른 유기체의 패스웨이와 그와 관련된 유전자 정보를 각각의 테이블에서 삭제하였다. 그리고 유전자와 관련된 GO 용어를 검색하기 위해 GO에서 제공하는 AmiGo[21] 테이블과 GO Annotation 테이블을 사용하여 18,195개 유전자 심볼을 매핑하였다. 매핑된 유전자 아이디를 기반으로 GO와 패스웨이 아이디를 추출하고, 그 외 관련된 GO와 패스웨이 정보를 추출한다. 또한 PRaDA 데이터베이스가 패스웨이 이미지를 저장하고 있기 때문에, 사용자 선택에 의해 패스웨이의 기본 정보뿐만 아니라 이미지도 제공된다.
질환 식별 모듈은 검색된 유전자 심볼 리스트를 통해 Online Mendelian Inheritance in Man (OMIM)[22] 데이터로부터 질환 정보를 검색하는 것으로, 유전자 식별기와 순차적으로, 패스웨이 식별기와는 병렬적으로 실행된다. OMIN에서 제공하는 테이블 형식은 하나의 질환에 여러 유전자 정보를 담고 있는 데이터 구조이므로 검색의 복잡함과 많은 검색 시간을 요구한다. 빠른 검색 기능을 필요로 하는 웹기반 서비스를 제공하여야하기 때문에 테이블을 유전자 기준으로 재구축하여 저장하였다. OMIN 테이블에는 3,637개 질환관련 아이디와 7,376개 유전자가 저장되어 있다. 세부적인 추가 정보를 제공하기 위해 PRaDA에서 OMIM 아이디를 선택하면 해당하는 관련 페이지가 링크된다.
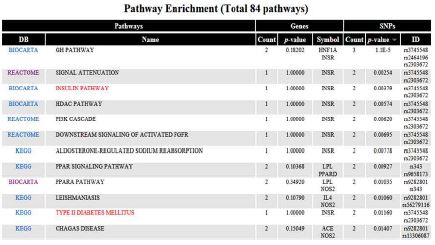
검색된 패스웨이와 단일염기다형성의 연관성을 분석하기 위해 유전자 집합 분석(GSEA)을 실행한다. 또한 유의한 단일염기다형성뿐만 아니라, 유의도가 낮은 다수의 단일염기다형성들의 집합이 간접적으로 질환 발병에 영향을 미칠 수 있기 때문에 단일염기다형성 집합 분석(SSEA)도 실행한다[5],[6], [8]. 패스웨이와 GO의 유의도 검사는 Fisher’s exact test를 변형한 EASE score를 이용하여 실행된다. 분석결과는 유의한 순서대로 패스웨이 이름, 유전자 리스트, 유전자 집합 분석의 유의도, 단일염기다형성 리스트, 단일염기다형성 집합 분석의 유의도를 나타내는 표 형식으로 웹 인터페이스에 제공된다.
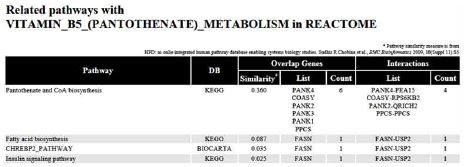
생물학적 패스웨이는 다수의 유전자 간 상호작용으로 구성되어 있고 순차적인 상호작용에 의해 발생하는 생물학적 기능의 단위이다. 유의하지 않은 단일염기다형성 집합들과 관련된 패스웨이의 변형은 기능적으로 근접한 또 다른 패스웨이의 변형을 일으킬 수 있다[8],[11],[23]. 그러므로 복합 질환은 단일 유전자의 산물 또는 단일 특정 패스웨이 분석만으로 원인 규명이 불가능하다. 본 논문에서는 더 넓은 질환 메카니즘을 이해하기 위해, 단일염기다형성이 위치하는 유전자가 참여하지 않는 패스웨이를 검색하고 그들의 간접적인 영향을 추정할 수 있는 근거를 제공한다.
단일염기다형성이 참여하는 패스웨이와 공통적으로 유전자를 공유하는 이웃 패스웨이를 근접 패스웨이 (realted pathway)라고 정의하였다. 일반적으로 패스웨이에 참여하는 유전자가 많을수록 근접 패스웨이 간 공유하는 유전자가 많을 것이다. 작은 규모의 패스웨이를 고려하여 근접 패스웨이는 패스웨이 간의 유사도[24]를 측정하여 검색한다. 유사도는 다음의 식(1)을 따른다.
| (1) |
N은 모든 패스웨이의 수이며, i번째와 j번째 패스웨이는 Pi와 Pj, |Pi|와 |Pj|는 패스웨이에 참여하는 유전자의 수이다. 패스웨이의 규모에 의해 발생하는 유사성 편향(bias)을 방지하기 위해, Human Pathway Database (HPD)[24]에서 제공하는 중량계수 (α, weight coefficient)와 작은 패스웨이의 유전자 수를 이용한다. 계산된 유사도가 Sij = - 1.01일 경우 패스웨이 i가 패스웨이 j의 소속된 하위 네트워크임을 의미하고, 반대의 경우 유사도 Sij = 1.01을 가진다.
웹 서비스의 특성상 입력된 단일염기다형성의 아이디 목록이 클 경우 전체 리스트를 화면에 표시하지 않고, p-value 기준 상위 100개의 단일염기다형성만 화면에 출력하게 된다. 따라서 검색된 단일염기다형성과 관련된 유전자, 패스웨이, 질환 정보는 사용자의 필요에 따라 선택적으로 텍스트 파일로 내려 받을 수 있다. 이 모듈은 서버에 파일을 생성하지 않고 http 프로토콜을 이용하여 직접 사용자의 클라이언트로 파일을 전송하는 방식을 사용한다.
Ⅲ. 결과 및 토론
PRaDA는 단일염기다형성 아이디를 기반으로 관련된 유전자와 패스웨이, 질환 정보를 검색하고, 통계적 분석을 실행하여 복합 정보를 제공하는 시스템이다 (Table 1). 두 가지 주요 기능의 첫 번째는 하나의 단일염기다형성 정보를 입력받아 단일염기다형성이 위치하는 모든 유전자와 패스웨이 정보를 순차적으로 검색해주는 기능이다. 사용자는 단계적으로 정보를 확인하며 관련 유전자나 패스웨이를 내부 데이터베이스 뿐만 아니라 외부경로를 통하여 확인할 수 있다. 두 번째는 다수의 단일염기다형성들이 미치는 복합적인 영향을 분석하기 위한 통계 분석이다. 사용자가 단일염기형성 리스트와 각각의 p-value 정보를 포함하는 PLINK[25] 출력물 형식의 텍스트 파일을 서버에 업로드 한다. 유의한 단일염기다형성을 선택하기 위해 p-value 임계치를 설정하면 분석할 단일염기다형성을 필터링하고 유전자, 패스웨이, 질환 정보를 검색하고 통계분석을 실행한다. 검색된 정보가 있는 단일염기다형성은 유의한 순서대로 인터페이스에 출력되고, 최종 결과물은 분석 정보를 모두 포함한 텍스트파일 형태로 다운로드가 가능하다.
이처럼 PRaDA는 기존의 패스웨이 분석 및 검색 도구들에 비해 더 다양한 패스웨이 데이터베이스를 정리하여 구축하였고, 그 데이터를 기반으로 단일염기다형성이 위치한 유전자의 생물학적 기능을 밝히고 그와 관련된 패스웨이와 질환 정보 및 통계 분석을 실행한다(Table 1). 업로드와 다운로드 기능을 추가하여 사용자가 이전 단계의 연구 결과를 분석하고 PRaDA의 결과를 다음 단계의 연구에 활용할 수 있도록 하였다. PRaDA의 종합적인 분석 결과는 질환이 발병하는 메카니즘을 이해하고 지표가 되는 유전자를 밝힐 수 있는 견해를 제공한다.
하지만 단일염기다형성의 영향으로 유전자의 발현 여부가 달라지기 때문에 패스웨이의 생물학적 영향이 질병이나 건강 상태에 따라 우리 몸에 다르게 발생할 수 있다. 따라서 실제 단일염기다형성에 의한 유전자 발현 차이와 그에 따라 달라지는 패스웨이 메카니즘을 질환 별로 이해하기 위해 전장유전체 연관분석 뿐만 아니라 전장전사체(RNA sequencing) 정보를 추가한 통합 연구가 필요하다.
References
-
P. Y. P. Kao, K. H. Leung, L. W. C. Chan, S. P. Yip, M. K. H. Yap, “Pathway analysis of complex diseases for GWAS, extending to consider rare variants, multi-omics and interactions”, Biochimica et Biophysica Acta, 1861(2), p335-353, (2017).
[https://doi.org/10.1016/j.bbagen.2016.11.030]

-
S. E. Kim, H. Kim, Y. Yun, S. G. Her, J. Cho, M. Kwon, Y. Chang, S. Ryu, H. Shin, C. Shin, N. H. Cho, Y. A. Sung, H. Kim, “Meta-analysis of genome-wide SNP- and pathway-based associations for facets of neuroticism”, Journal of Human Genetics, 62, p903-909, (2017).
[https://doi.org/10.1038/jhg.2017.61]

-
J. Wu, X. Mao, T. Cai, J. Luo, L. Wei, “KOBAS server: a web-based platform for automated annotation and pathway identification”, Nucleic Acids Research, Vol. 34, W720-724, 2006
[https://doi.org/10.1093/nar/gkl167]

-
L. Weng, F. Macciardi, A. Subramanian, G. Guffanti, S. G. Potkin, Yu Z., X. Xie, “SNP-based pathway enrichment analysis for genome-wide association studies”, BMC Bioinformatics, 12, 99, (2011).
[https://doi.org/10.1186/1471-2105-12-99]

-
I. Medina, D. Montaner, N. Bonifaci, M. A. Pujana, J. Carbonell, J. Tarraga, J. Dopazo, “Gene set-based analysis of polymorphisms: finding pathways or biological processes associated to traits in genome-wide association studies”, Nucleic Acids Research, 37, pW340-344, (2009).
[https://doi.org/10.1093/nar/gkp481]

-
P. Holmans, E. K. Green, J. S. Pahwa, M. A. Ferreira, S. M. Purcell, P. Sklar, Wellcome Trust Case-Control ConsortiumM. J. Owen, M. C. O'Donovan, N. Craddock, “Gene ontology analysis of GWA study data sets provides insights into the biology of bipolar disorder”, American Journal of Human Genetics, 85, p13-24, (2009).
[https://doi.org/10.1016/j.ajhg.2009.05.011]

-
D. Zamar, B. Tripp, G. Ellis, D. Daley, “Path: a tool to facilitate pathway-based genetic association analysis“, Bioinformatics, 25, p2444-2446, (2009).
[https://doi.org/10.1093/bioinformatics/btp431]

-
R. M. Cantor, K. Lange, J. S. Sinsheimer, “Prioritizing GWAS Results: A review of statistical methods and recommendations for their application“, American Journal of Human Genetics, 86, p6-22, (2010).
[https://doi.org/10.1016/j.ajhg.2009.11.017]

-
K. Zhang, S. Cui, S. Chang, L. Zhang, J. Wang, “i-GSEA4GWAS: a web server for identification of pathways/gene sets associated with traits by applying an improved gene set enrichment analysis to genome-wide association study“, Nucleic Acids Research, Vol. 38, W90-95, 2010
[https://doi.org/10.1093/nar/gkq324]

-
E. Cirillo, M. Kutmon, M. G. Hernandez, T. Hooimeijer, M. E. Adriaens, L. M. T. Eijssen, L. D. Parnell, S. L. Coort, C. T. Evelo, “From SNPs to pathways: Biological interpretation of type 2 diabetes (T2DM) genome wide association study (GWAS) results”, PLoS ONE, 13(4), (2018).
[https://doi.org/10.1371/journal.pone.0193515]

-
D. W. Huang, B. T. Sherman, R. A. Lempicki, “Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists“, Nucleic Acids Res, 37, p1-13, (2009).
[https://doi.org/10.1093/nar/gkn923]

-
G. Peng, H. Luo, H. Siu, Y. Zhu, P. Hu, J. Zhao, X. Zhou, J. D. Reveille, L. Jin, C. I. Amos, M. Xiong, “Gene and pathway-based second-wave analysis of genome-wide association studies“, European Journal of Human Genetics, 18, p111-117, (2010).
[https://doi.org/10.1038/ejhg.2009.115]

-
H. J. Ban, J. Y. Heo, K. S. Oh, K. J. Park, “Identification of type 2 diabetes-associated combination of SNPs using support vector machine“, BMC Genetics, 23, p11-26, (2010).
[https://doi.org/10.1186/1471-2156-11-26]

-
K. Zhang, S. Chang, S. Cui, L. Guo, L. Zhang, J. Wang, “ICSNPathway: identify candidate causal SNPs and pathways from genome-wide association study by one analytical framework”, Nucleic Acids Research, 39, pW437-443, (2011).
[https://doi.org/10.1093/nar/gkr391]

-
D. F. Schwarz, O. Hädicke, J. Erdmann, A. Ziegler, D. Bayer, S. Möller, “SNPtoGO: characterizing SNPs by enriched GO terms“, Bioinformatics, 24, p146-148, (2008).
[https://doi.org/10.1093/bioinformatics/btm551]

- dbSNP : a database of single nucleotide polymorphisms [Internet]. Available: http://www.ncbi.nlm.nih.gov/projects/SNP
- refGene [Internet]. Available: http://www.ncbi.nlm.nih.gov/RefSeq
-
K. Sidiropoulos, G. Viteri, C. Sevilla, S. Jupe, M. Webber, M. Orlic-Milacic, B. Jassal, B. May, V. Shamovsky, C. Duenas, K. Rothfels, L. Matthews, H. Song, L. Stein, R. Haw, P. D’Eustachio, P. Ping, H. Hermjakob, A. Fabregat, “Reactome enhanced pathway visualization”, Bioinformatics, 33(21), p3461-3467, (2017).
[https://doi.org/10.1093/bioinformatics/btx441]

-
A. Subramanian, P. Tamayo, V. K. Mootha, S. Mukherjee, B. L. Ebert, M. A. Gillette, A. Paulovich, S. L. Pomeroy, T. R. Golub, E. S. Lander, J. P. Mesirov, “Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles“, Proc Natl Acad Sci USA,, 102, p15545-15550, (2005).
[https://doi.org/10.1073/pnas.0506580102]

- Kyoto Encyclopedia of Genes and Genomes (KEGG) [Internet]. Available: http://www.genome.jp/kegg
- Gene Ontology database [Internet]. Available: http://www.geneontology.org
- A. Hamosh, A. F. Scott, J. S. Amberger, C. A. Bocchini, V. A. McKusick, “Online Mendelian Inheritance in Man (OMIM), a knowledgebase of human genes and genetic disorders”, Nucleic Acids Research, 33(Suppl._1), pD514-D517, (2005).
-
M. A. Garcia-Campos, J. Espinal-Enriquez, E. Hernandez-Lemus, “Pathway Analysis: State of the Art”, Frontiers in Physiology, 6, p383, (2015).
[https://doi.org/10.3389/fphys.2015.00383]

-
S. R. Chowbina, X. Wu, F. Zhang, P. M. Li, R. Pandey, H. N. Kasamsetty, J. Y. Chen, “HPD: an online integrated human pathway database enabling systems biology studies“, BMC Bioinformatics, 10(suppl 11), pS5, (2009).
[https://doi.org/10.1186/1471-2105-10-S11-S5]

-
S. Purcell, B. Neale, K. Todd-Brown, L. Thomas, M. A. R. Ferreira, D. Bender, J. Maller, P. Sklar, P. I. W. de Bakker, M. J. Daly, P. C. Sham, “PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses”, The American Journal of Human Genetics, 81(3), p559-575, (2007).
[https://doi.org/10.1086/519795]


유기진(Kijin Yu)
2006년 : 충북대학교 생물학과 (이학사)
2008년 : 충북대학교 대학원 전자계산학과 (공학석사)
2013년 : 충북대학교 대학원 컴퓨터과학과 (공학박사 수료)
2008년~2010년: 한국생명공학연구원
2010년~2013년: 질병관리본부 국립보건연구원
2017년~현 재: 아산생명과학연구원 근무 중
※관심분야: 바이오인포매틱스, 바이오메디칼, 데이터마이닝, 데이터베이스

박수호(Soo Ho Park)
2013년 : 충북대학교 대학원 컴퓨터과학과 (공학석사)
2015년 : 충북대학교 대학원 컴퓨터과학과 (공학박사 수료)
2016년~2016년: 엔셀주식회사
2016년~현 재: 주식회사엔젠바이오 근무 중
※관심분야: 데이터마이닝, 데이터베이스, 바이오메디칼, 바이오인포매틱스

류근호(Keun Ho Ryu)
1976년 : 숭실대학교 전산과 (공학사)
1980년 : 연세대학교 공학대학원 전산전공 (공학석사)
1988년 : 연세대학교 대학원 전산전공 (공학박사)
2012년 : 몽고 국립대 University of Mongolia (명예박사)
1976년~1986년: 육군군수 지원사 전산실(ROTC 장교), 한국전자통신연구원(연구원), 한국방송통신대학교 전산학과(조교수)
1989년~1991년: Univ. of Arizona Research Staff
1986년~현 재: 충북대학교 소프트웨어학과 교수
※관심분야: 시간 데이터베이스, 시공간 데이터베이스, Temporal GIS, 지식기반 정보검색 시스템, 유비쿼터스 컴퓨팅 및 스트림데이터 처리, 데이터 마이닝, 데이터베이스 보안, 바이오 인포메틱스 및 바이오메디칼