혼합적 속성관계에 기반한 의료영상 분류
Copyright ⓒ 2020 The Digital Contents Society
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-CommercialLicense(http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.

초록
의료영상시스템은 정보기술을 이용하여 빠르게 발전하고 있다. 대다수 병원에서 의료영상시스템을 사용하며, 의료영상시스템은 의료영상의 저장과 전송에 이용된다. 일반적으로 의료영상 분류에서 사용하는 분류방식은 주석처리방식과 내용처리방식이다. 주석처리방식은 전문가 의견을 포함하기 때문에 우수한 분류 결과를 생성하나, 시스템 구축에 많은 시간과 비용이 필요하다. 내용처리방식은 의료영상시스템을 자동화할 수 있는 장점이 있으나, 의료영상에서 내용기반 속성을 추출하기는 매우 어렵다. 본 논문에서 혼합적 속성관계를 이용한 의료영상 분류기법을 제안한다. 혼합적 속성관계에 기반한 분류기법의 우수성을 실험으로 확인하였다.
Abstract
Medical imaging systems have been rapidly advancing using information technology. Most medical centers use the medical imaging systems. Medical imaging systems are used to store and transfer medical images. Medical images are generally classified using the comment and contents processing methods. The comment processing method generates excellent results because it contains professional opinions. However, the comment processing method requires substantial time and costs for effective system implementation. The contents processing method is effective in automating the medical imaging system. However, it is difficult to extract contents-based attributes from medical images. In this paper, we propose the classification method of medical images based on mixed attributes relation. The superiority of the classification method based on mixed attributes relation was verified through the experiment.
Keywords:
Medical Image, Image Classification, Machine Learning, Brain Tumor, MRI키워드:
의료영상, 영상분류, 기계학습, 뇌종양, 자기공명영상Ⅰ. 서 론
의료 정보화는 정보기술을 바탕으로 빠르게 발전하고 있으며, 의료 정보화는 전자의무기록(EMR)이나 의료영상저장전송시스템(PACS) 등을 중심으로 큰 발전을 이루고 있다. 다양한 형태로 존재하는 의료영상저장전송시스템은 3차 의료기관을 중심으로 퍼져나갔으며, 최근에는 하위 의료기관까지 의료영상저장전송시스템을 활용하고 있다. 의료영상저장전송시스템은 네트워크를 이용하여 의료영상을 전달하는 시스템이다. 의료영상저장전송시스템을 이용함으로써 의료진들은 빠르고 쉽게 의료업무를 진행할 수 있다.
의료영상저장전송시스템은 환자의 의료영상을 활용하여 질환을 진단하고 예측할 때 활용되는 의료활동 보조도구이다. 의료영상저장전송시스템에서 활용하는 의료영상 이미지는 MRI와 CT가 있으며, 다른 의료영상 이미지로는 X-ray가 있다[1]. 의료활동 보조도구로 활용되는 의료영상저장전송시스템은 그레이스케일(Gray-scale)로 표현된 의료영상을 활용한다[2]-[3]. X-ray는 해상도가 낮으나 가격이 저렴하다는 장점이 있고, MRI는 다른 의료영상에 비해 해상도가 높으나 가격이 비싸다는 단점이 있다[4].
의료진의 의료활동에 보조적 수단으로 사용하는 의료영상저장전송시스템은 일반적으로 획득된 의료영상을 저장하고 전송하는 일을 수행한다. 의료영상저장전송시스템은 필름을 가지고 의료활동에 사용하는 과거 방법을 탈피하고 의료영상 이미지를 디지털 방식으로 변환하여 사용함으로써 의료 정보화에 크게 기여하고 있다. 의료영상저장전송시스템의 도입으로 의료정보에 대한 자동화와 정보화는 빠르게 성장하였으며, 이러한 의료정보의 자동화와 정보화는 의료진의 의료활동에 편리성을 제공하고 업무시간을 단축하였다.
의료영상저장전송시스템은 이러한 의료 정보화를 기반으로 많은 발전은 이루었으나 영상정보와 진단정보가 분리된 경우가 많다. 현재 상황에서 영상정보와 진단정보의 정보화는 의료진의 적극적 개입을 요구하는 경우가 빈번하게 발생하기 때문에 많은 업무가 발생하는 의료진에게 이러한 의료업무에 대한 적극적 개입은 의료업무에 상당히 큰 부담일 수 있다. 이러한 문제점을 해결하기 위해 의료영상저장전송시스템은 의료진의 의료업무를 줄이는 방법을 제공해야 한다.
일반적으로 의료영상시스템은 한 명의 환자를 촬영하여도 다수의 의료영상이 발생하며, 높은 해상도와 다수의 의료영상 데이터는 큰 저장공간을 요구한다. 이러한 의료영상의 특성은 의료영상 분류에서 단점으로 작용한다. 의료영상의 형태적 특성으로 볼 때 그레이스케일에 기반한 의료영상의 단순성은 의료영상의 특징 추출에 어려움으로 작용한다[5]. 이러한 그레이스케일이라는 색의 특성은 의료영상의 분류에 문제를 발생시킬 수 있으며, 그레이스케일의 형식적 단순성은 질환의 특성을 명확히 나타내기 어렵다는 이차적 문제도 발생시킨다.
의료영상의 분류에 활용되는 방식의 하나인 주석처리방식은 전문가들의 개입을 요구하는 방식이다. 주석처리방식은 의료영상에 대한 진단정보나 질환의 주석을 제공함으로써 안정된 의료정보를 제공하는 방식이다[6]. 주석처리방식은 해당 분야 전문가의 직접적 분석을 통해 의료정보를 제공하기 때문에 의료영상 분석에 매우 효과적이다. 그러나 이러한 주석처리방식은 전문가의 적극적 개입이 필요하여 의료시스템 구축에 많은 시간과 비용이 필요하다는 단점이 있다. 다른 방식으로는 내용처리방식이 있으며, 내용처리방식은 의료영상의 형태적 특성을 이용하는 의료영상을 분석하는 방식으로 주석처리방식보다 시간과 비용이 적게 사용된다는 장점이 있다[7]. 내용처리방식에서 사용하는 기본적인 의료영상 속성은 질감이나 모양 등이다. 내용처리방식은 의료영상의 분류에서 자동화를 구축할 수 있는 우수한 도구이다[8].
의료영상의 특징인 형식적 단순성과 다수로 존재하는 의료영상의 크기 문제를 해결하고 의료영상의 분류에 효과적인 방식은 속성 혼합이다. 다양한 특성을 나타내지 못하는 의료영상의 단점을 해결하기 위해 의료영상에서 추출한 기본적 속성을 해부학적 단위객체와 혼합하여 새로운 속성을 생성하면 다양한 속성을 생성할 수 있다. 이러한 속성의 다양성은 그레이스케일 방식의 단순성을 해결하고 의료영상의 분류 자동화에 효과적으로 사용할 수 있다.
본 논문에서 제안한 혼합적 속성관계를 이용한 의료영상 분류기법은 의료영상에 포함된 해부학적 단위객체들의 공간적 특성을 고려하여 단위객체의 관계성을 추출하고, 의료영상에 포함된 기본적 속성들과 단위객체와의 관계성을 혼합하여 혼합적 속성관계를 생성한다. 혼합적 속성관계를 의료영상의 분류에 적용하면 의료영상의 단순성을 극복하여 의료영상 분류에 효과적으로 사용할 수 있는 장점이 있다.
Ⅱ. 의료영상의 특징과 검색
의료영상은 신경학적검사(Neurologic examination)나 이학적검사(Physical examination) 등에 활용한다. 의료영상은 일반적인 영상과 큰 차이가 있으며, 흑백 이미지로 존재한다. 흑백 이미지는 유색 이미지보다 질감 특징이 잘 표현되지 않으며, 객체의 영역을 구분하기도 어렵다[9].
활용도 높은 의료영상으로 X-ray가 있으며, X-ray는 저렴하다는 장점은 있으나 해부학적 단위객체의 구분이 명확하지 않다는 단점이 있다. X-ray는 X선을 통해 신체 내부를 촬영하여 얻어진 영상으로 해부학적 구조를 분석하는 데 이용하며, 골격이나 흉부 또는 복부 등을 촬영할 때 주로 활용한다. X-ray는 신체 촬영에 X선을 사용한다는 단점이 있지만, 가격이 저렴하다는 장점이 있다. X-ray 영상을 이용한 의료영상시스템에서 자주 활용하는 하나의 방법은 히스토그램이다. 히스토그램은 연속적 구간을 임의의 단위 구간으로 나누어 데이터의 출현을 분석하는 방법이다[10]. X-ray의 결과물인 의료영상에 히스토그램을 활용하면 의료영상의 전체적 구조를 쉽게 파악할 수 있다는 장점은 있으나, X-ray의 해상도 문제로 분석이 어려울 수 있다는 단점이 있다. 그래서 추가적인 분석 수단으로 해부학적 단위객체의 크기나 윤곽선 등을 활용하기도 하나, 여기에서도 X-ray의 해상도 문제는 여전히 존재한다.
MRI는 의료영상에서 가장 높은 해상도를 나타내어 질환을 분석하는 보조적 도구로는 매우 우수한 효과를 나타내지만, 비용이 많이 든다는 단점이 있다. MRI는 자력을 이용하여 신체의 내부구조를 촬영하며, 근육이나 혈관 등도 촬영할 수 있다. MRI는 방사선을 사용하지 않기 때문에 인체에 나쁜 영향을 미치는 요소가 발생하지 않는다는 장점이 있다[11]-[12]. 촬영 방법에 따라 다양한 각도에서 의료영상을 생성할 수 있으며, 뇌종양이나 뇌출혈을 진단할 때 매우 효과적이다. MRI는 해상도가 높아 히스토그램을 활용하여도 다른 의료영상보다 우수한 분석률을 나타낸다. 그러나 MRI도 그레이스케일에 기반한 의료영상이기 때문에 히스토그램만 활용하면 의료영상의 분류에 한계가 있다[13]. 이러한 문제점을 해결하기 위해 MRI를 이용한 분류시스템은 의료영상에 포함된 해부학적 단위객체의 모양이나 크기 등을 이용하여 의료영상 분류시스템에 사용한다[14]. 그러나 MRI도 그레이스케일에 기반한 의료영상이기 때문에 기본적으로 의료영상의 형태학적 구조가 단순하다는 단점이 있다[15].
의료영상시스템의 분류에 활용하는 주석처리방식은 전문가가 의견을 직접 작성하는 방식을 취함으로써 의료영상 분류에 대한 정확도가 상대적으로 높다는 장점이 있다. 그러나 전문가의 직접적 개입은 시스템 구축에 많은 시간과 비용을 소모한다는 단점이 있다. 그리고 전문가들이 의료영상에 주석을 처리할 때 통일된 규칙이 없으므로 정보를 일반화하기 어렵다는 단점을 내포한다. 주석처리에 대한 다양한 표현방식은 의료영상 분류 자동화에 악영향을 미치어 의료영상 자동 분류시스템에 주석처리방식을 적용할 경우, 키워드와 같은 이차적 속성을 생성해야 하는 부가적 작업이 필요하다.
내용처리방식은 의료영상에서 추출할 수 있는 질감이나 모양 등과 같은 형태학적 특성 등을 활용하여 해부학적 단위객체의 속성을 생성한다. 내용처리방식은 의료영상의 해부학적 단위객체 속성들을 추출하여 일반화된 표현방식에 따라 의료영상시스템의 자동화에 적용한다. 이러한 표준화된 방식은 의료영상시스템의 자동화에 효과적으로 적용할 수 있다. 그러나 내용처리방식은 의료영상의 표면적 질감이나 형상적 구조를 활용하기 때문에 다양한 속성정보를 생성하기 어렵다는 단점이 있다[16]. 내용처리방식은 그레이스케일에 기반한 의료영상을 활용하여 해부학적 단위객체를 추출하는 것부터 의료영상 속성을 생성하는 것까지 자동화가 가능하다. 이러한 구조는 시간과 비용적 측면에서 시스템 구축에 효과적이기는 하나, 의료영상 속성의 일반화와 단순성은 의료영상시스템의 분류 정확도에 나쁜 영향을 미친다. 내용처리방식은 의료영상의 표면정보를 활용하기 때문에 영상의 의미를 분석하기는 매우 어렵다. 이러한 문제로 동일한 해부학적 단위객체들이 서로 다른 모양으로 존재한다면 의료영상의 분류 정확도는 낮아지게 될 것이다.
본 논문에서 이러한 내용처리기법의 문제점을 해결하기 위해 혼합적 속성관계기법을 제안한다. 혼합적 속성관계기법은 의료영상 단위객체의 단순성을 해결하고, 의료영상의 빈약한 속성정보를 풍부하게 만들어 의료영상시스템의 성능을 우수하게 확보할 수 있다는 장점이 있다. 혼합적 속성관계기법은 의료영상에서 해부학적 단위객체의 고유 특징을 추출하고, 단위객체의 공간적 특성을 나타내는 속성집합과 연결성을 측정하여 해부학적 단위객체의 이차속성을 생성함으로써 의료영상의 속성정보를 풍부하게 만드는 장점이 있다.
Ⅲ. 혼합적 속성 관계
3-1 속성 관계의 표현
의료영상을 분류하기 위해서는 의료영상에 포함된 특징 속성이나 해부학적 단위객체들을 추출해야 한다. 의료영상에 포함된 속성들은 단일한 형식이 아니라 다양한 형식과 구조로 존재하며, 의료영상에서 추출할 수 있는 대표적 속성은 색상과 형태 그리고 질감 등이다.
의료영상에서 추출할 수 있는 다양한 형식의 속성들을 혼합적 속성관계기법에서는 상위집합과 하위집합으로 분류하여 의료영상시스템에 사용한다. 상위집합과 하위집합에 대한 속성들을 표 1에 나타냈다. 하위집합은 해부학적 단위객체의 중심, 단위객체의 밝기, 단위객체의 길이, 단위객체의 면적 등을 포함한다. 하위집합에 포함된 정보는 해부학적 단위객체의 밝기나 길이 등을 포함한다. 하위집합을 활용하면 전문가의 적극적 개입이 없어도 의료영상시스템을 자동화할 수 있다는 장점이 있다. 상위집합은 의료영상에서 추출한 해부학적 단위객체의 해부학적 정보와 단위객체의 관계성을 포함한다. 상위집합에는 의료영상에서 추출한 단위객체들의 모양과 서로 다른 단위객체의 중첩도 등을 포함한다. 상위집합은 해부학적 단위객체의 원형도, 단위객체의 사각도, 단위객체의 돌출도, 단위객체의 중첩도 등을 포함한다.
의료영상 분류기에서 사용하는 의료영상은 해부학적 특성에 따라 단위객체로 분할된다. 분할된 해부학적 단위객체는 백질이나 회백질 그리고 뇌척수액 등이며, 해부학적 단위객체들의 속성은 상위집합과 하위집합과 포함된다. 단위객체의 길이는 픽셀의 값을 활용하며, 단위객체의 밝기는 색상 히스토그램을 활용한다. 단위객체의 돌출도는 단위객체의 비원형율을 표현하고, 단위객체의 원형도는 해부학적 단위객체의 평균 곡률을 표현한다.
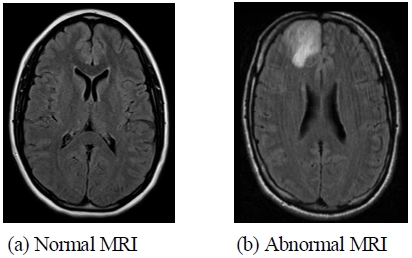
그림 1에 MRI 영상 샘플을 나타냈다. 좌측 MRI는 정상 소견에 대한 MRI 영상이고, 우측 MRI는 병적 소견이 나타난 MRI 영상이다.
3-2 단위객체의 군집화
해부학적 단위객체는 혼합적 속성관계를 이용하여 특성을 재표현하며, 원형도는 식 1과 같다. 식1에서 X와 Y는 의료영상의 해부학적 단위객체의 외곽경계에 해당하는 좌표집합이다. 외곽경계 좌표집합에서 동일한 벡터에 존재하는 xi와 yi 성분은 해부학적 단위객체에서 개별적 원형을 생성한다. 혼합적 속성관계기법은 이러한 단위객체에 소속된 벡터 성분을 이용하여 개별적 벡터기반 원형을 생성하고, 생성된 개별적 원형은 벡터기반 원형집합에 포함된다. 벡터기반 원형집합은 식 1에 의해 평균 원형도를 측정한다.
| (1) |
의료영상에서 추출된 종양 객체는 다른 의료영상 객체에 비해 원형도가 1에 가깝다. 일반적으로 종양은 방사형으로 진행하는 특성이 있어 다른 해부학적 객체와는 다르게 원형도가 높다는 특징이 있다. 그러나 종양의 침윤이 발생하면 다른 양상을 나타낸다. 그래서 본 논문에서는 이러한 종양의 특징을 특화하기 위해 원형도를 사용하여 의료영상 분류에 적용한다. 원형집합에 포함된 다양한 크기의 원형들은 해부학적 단위객체 특성을 나타낸다. 뇌척수액은 악성 종양에 비해 원형도가 높기 때문에 원형집합의 카디낼리티(Cadinality)가 높다는 특성이 있다.
해부학적 단위객체 정보를 포함한 원형집합을 활용하여 혼합적 속성관계기법은 최소값을 소유한 벡터기반 원형의 크기를 추출함으로써 단위객체에 대한 점유도집합을 생성한다. 해부학적 단위객체의 최소 점유도를 추출하는 방식은 식 2와 같다. 공식 2에서 Cij 와Cop는 해부학적 단위객체의 원형집합에 속한 속성들이다. xi와 yi 성분은 해부학적 단위객체의 혼합적 속성을 연속적으로 생성한 좌표 속성에 해당한다. 각 좌표 속성은 벡터기반 원형을 만들고 식 2에 의해 점유도를 추출한다.
| (2) |
원형집합에 속하는 해부학적 단위객체의 원형에서 가장 큰 점유도를 생성할 때는 식 3을 이용한다. 점유도집합은 내부 점유도집합과 외부 점유도집합으로 나누며, 내부 점유도집합에는 해부학적 단위객체의 최소 크기에 대한 점유도 속성이 포함되고, 외부 점유도집합에는 해부학적 단위객체의 최대 크기에 대한 점유도 속성이 포함된다.
| (3) |
해부학적 단위객체들을 군집화하기 위해서는 각 단위객체들의 거리를 구해야 한다. 다차원 공간상에서 연속적 특성을 소유한 객체들의 거리를 구하기 위해서는 이산적 속성을 생성해야 한다.
| (4) |
해부학적 단위객체의 거리를 계산할 때 적용되는 방식은 식 4와 같다. Ri와 Rj는 혼합적 속성으로 변환된 해부학적 단위객체들의 원형도 속성을 의미한다. xi와 yi는 해부학적 단위객체의 혼합적 속성이 적용된 단위객체 R 속성의 i번째 원형도 중심거리 생성 요소이다. 해부학적 단위객체의 거리함수는 혼합적 속성으로 변환된 단위객체들의 거리를 측정하는 함수이며, 거리함수에 의해 의료영상에 포함된 해부학적 단위객체들은 집단화에 적용된다.
Ⅳ. 실험결과
4-1 실험환경
실험에 사용한 의료영상 데이터 집합은 크게 두 가지이다. 두 개의 데이터 집합을 실험에 활용한 이유는 의료영상 데이터는 일반적으로 전문가 소견에 의해 크게 두 가지로 나뉜다. 하나는 질환이 나타나지 않은 정상 소견 의료영상이고, 다른 하나는 질환에 대한 소견이 나타난 의료영상이다. N_imagesSet 데이터 집합은 정상적인 소견에 대한 의료영상 데이터 집합이고, A_imagesSet 데이터 집합은 병적 소견에 대한 의료영상 데이터 집합이다. 병적 소견을 나타낸 의료영상에는 두개인두종과 혈관모세포종과 같은 질환이 나타나 있다. 실험에 사용한 데이터 집합은 크기로 분류될 수 있으며, 크게 두 개의 데이터 집합으로 나뉜다. Sset 데이터 집합은 150개의 의료영상 데이터를 포함하고 있고, Lset 데이터 집합은 의료영상 데이터를 500개 포함하고 있는 가장 큰 데이터 집합이다.
실험환경은 1차 실험환경과 2차 실험환경으로 나뉜다. 1차 실험환경에서는 Tumor_I 데이터 집합을 활용하고, 2차 실험환경에서는 Tumor_II 데이터 집합을 활용한다. Tumor_I은 두개인두종에 관련한 실험 데이터 집합이고, Tumor_II는 혈관모세포종에 관련한 실험 데이터 집합이다. 혈관모세포종의 일반적 발생 위치는 소뇌 부위이며, 형태학적 위치로 대뇌의 하부에 나타나는 경우가 많다. 그리고 혈관모세포종이 발생한 의료영상에서는 뇌척수액의 해부학적 단위객체가 잘 나타나지 않는다는 특징이 있다. 혈관모세포종은 다른 해부학적 단위객체들보다 크기가 작다. 혈관모세포종의 발생 위치는 뇌의 중심점을 기준으로 뒤쪽에 나타난다. 이러한 형태학적 정보를 이용하면 해부학적 단위객체의 위치나 크기가 의료영상 분류에 중요한 요소로 작용한다. 두개인두종에 대한 해부학적 단위객체의 일반적 발생 위치는 뇌하수체 주변이다. 두개인두종의 형태학적 위치는 뇌하수체의 하부에 존재하는 경우가 많아 뇌척수액에 해당하는 단위객체가 잘 나타나지 않는다. 빈발 부위는 대뇌 밑에 속하고 두개인두종의 크기는 작게 나타난다는 특징이 있다.
실험에 사용된 의료영상 분류기법은 두 가지 타입으로 Method_A와 Method_B이다. Method_A는 거리기반 군집화기법으로 해부학적 단위객체의 중심점을 기반으로 각 단위객체의 유사성을 측정하는 방법을 따른다. 일반적으로 내용기반 분류방식에서 많이 활용하는 기법으로 객체들의 거리를 이용하기 때문에 분류 정확도가 높은 기법이다. Method_B는 본 논문에서 제안한 혼합적 속성관계기법이다.
4-2 실험분석
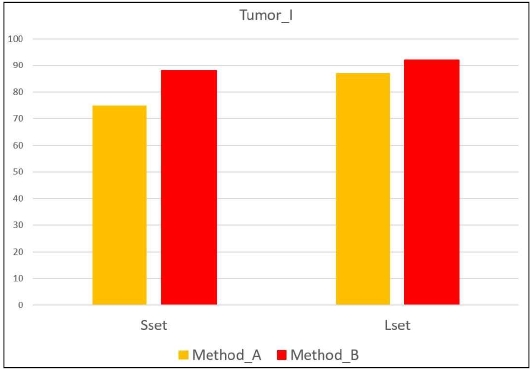
Tumor_I 실험환경에서의 실험결과를 그림 2에 나타냈다. 거리기반 군집화기법은 Sset 데이터 집합에서 75%의 성능을 나타내었고, 혼합적 속성관계기법은 88%의 성능을 나타냈다. Sset 데이터 집합은 소용량 의료영상 데이터 집합으로 적은 수량의 해부학적 단위객체 정보를 포함하고 있다. 소용량의 단위객체 정보를 포함하고 있는 데이터 집합에서 혼합적 속성관계기법은 해부학적 단위객체의 혼합적 속성관계를 분류에 적용함으로써 거리기반 군집화기법에 비해 13% 높은 성능을 나타내었다. 거리기반 군집화기법은 해부학적 단위객체의 속성에 대한 거리를 활용하여 군집화를 수행하기 때문에 다양한 서로 다른 속성들이 존재하는 데이터 집합에서는 우수한 성능을 나타내지 못하였다.
Lset 데이터 집합에서는 거리기반 군집화기법이 88%의 성능을 나타내어 Sset에서의 성능보다 12% 높은 성능을 나타내었다. 거리기반 군집화기법이 Lset에서 우수한 성능을 나타내는 이유는 해부학적 단위객체의 수량이 증가함에 따라 속성정보 수량도 같이 증가하기 때문이다. 의료영상 단위객체 정보가 증가하면 속성들의 복잡도가 낮아지는 특성이 있다. 이러한 복잡도 감소는 거리기반 군집화기법이 유사 단위객체를 추출할 때 의료영상의 분류 성능을 향상하는 요인으로 작용한다.
혼합적 속성관계기법은 Lset 데이터 집합에서 92%의 성능을 나타내어 거리기반 군집화기법보다 5% 높은 성능을 나타냈다. 혼합적 속성관계기법은 해부학적 단위객체의 속성 관계성을 이용하여 의료영상 분류를 수행하기 때문에 데이터의 크기 증가에 따라 지속적인 성능 향상을 나타냈다. 소규모 데이터 집합보다 대규모 데이터 집합에서 낮은 성능 향상을 나타낸 이유는 두개인두종의 해부학적 단위객체 특성이 다른 단위객체보다 분류에 효과적인 공간적 특성이 있기 때문이다. 이러한 이유에서 Lset 데이터 집합에서 혼합적 속성관계기법의 성능이 Sset 데이터 집합에서의 성능보다 4% 높은 낮은 성능 향상을 보였다.
이렇게 낮은 성능 향상이 나타난 이유는 다른 해부학적 단위객체에 비해 Tumor_I에 속한 단위객체들은 속성 특성이 분류에 효과적이기 때문이다. 이러한 두개인두종의 특성으로 혼합적 속성관계기법이 소용량 데이터에서도 해부학적 단위객체의 특성을 잘 추출하였기 때문에 대용량 데이터에서 높은 성능 향상을 나타내지 않은 것이다.
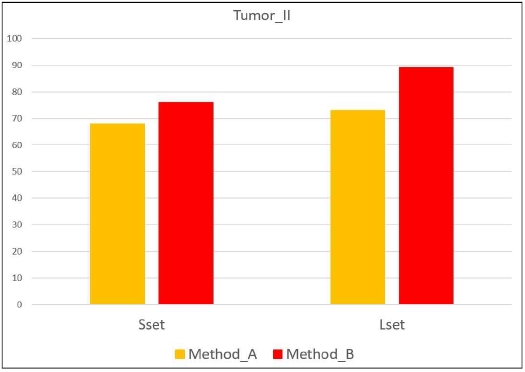
Tumor_II 실험환경에서의 실험결과를 그림 3에 나타냈다. 혼합적 속성관계기법은 Sset 데이터 집합에서 76%의 성능을 나타냈고, 거리기반 군집화기법은 68%의 성능을 나타냈다. 소용량 데이터 집합에서 혼합적 속성관계기법은 거리기반 군집화기법보다 8% 높은 성능을 나타냈다. 2차 실험환경은 1차 실험환경보다 해부학적 단위객체의 복잡도가 높은 데이터 집합들을 포함하고 있어 2차 실험환경이 1차 실험환경에 비해 낮은 성능을 나타냈다. Lset 데이터 집합에서 혼합적 속성관계기법은 89%의 성능을 나타냈고, 거리기반 군집화기법은 73%의 성능을 나타냈다. 소용량 데이터 집합보다 대용량 데이터 집합에서 혼합적 속성관계기법이 거리기반 군집화기법보다 높은 성능을 나타낸 이유는 혼합적 속성관계기법은 속성의 관계성을 활용하여 해부학적 단위객체의 복잡도를 낮추기 때문이다.
2차 실험환경은 1차 실험환경보다 복잡도가 높은 데이터 집합을 활용하고 있어 1차 실험환경보다 2차 실험환경에서 혼합적 속성관계기법과 거리기반 군집화기법 모두 낮은 성능을 나타냈다. 그러나 혼합적 속성관계기법은 복잡도가 높은 데이터 집합에서도 데이터의 증가에 따라 빠른 성능 향상을 나타냈지만, 거리기반 군집화기법은 복잡도가 높은 데이터 집합에서 데이터가 증가하더라도 높은 성능 향상을 나타내지 못하였다. 이러한 실험결과만 보더라도 혼합적 속성관계기법은 해부학적 단위객체의 복잡도가 증가하더라도 안정된 성능 향상을 나타낼 수 있다는 것을 실험을 통해 확인하였다.
Ⅴ. 결 론
의료영상에 포함된 해부학적 단위객체는 내용기반 속성을 포함하고 있으나, 속성정보가 풍부하지 않아 의료영상 분류에 효과적인 방법을 제시하기 어렵다. 이러한 문제점을 고려하여 본 논문에서 제안한 혼합적 속성관계기법은 의료영상에 포함된 해부학적 단위객체들의 공간적 특성을 고려하여 혼합적 속성관계를 생성한다. 혼합적 속성관계기법은 하위집합의 속성과 상위집합의 속성에 대한 연결성을 측정하여 해부학적 단위객체의 새로운 특성을 추출한다. 혼합적 속성관계기법을 의료영상의 분류에 활용하면 효과적으로 의료영상을 분류할 수 있다는 장점이 있다.
혼합적 속성관계기법은 의료영상에 포함된 해부학적 단위객체의 공간적 특성을 의료영상 분류에 이용함으로써 해부학적 단위객체의 복잡도를 낮추는 특성이 있다. 이러한 이유에서 혼합적 속성관계기법은 복잡도가 높은 데이터 집합에서도 안정된 성능을 유지할 수 있다. 상대적으로 거리기반 군집화기법은 데이터의 복잡도가 증가함에 따라 불안정한 성능을 나타냈다. 거리기반 군집화기법은 의료영상 단위객체의 형태학적 특성을 이용하기 때문에 복잡도가 낮은 데이터 집합에서 데이터가 증가하면 높은 성능 향상을 나타내지만, 복잡도가 높은 데이터 집합에서는 해부학적 단위객체의 특성이 잘 나타나지 않아 불안정한 성능을 나타냈다.
본 논문에서 제안한 혼합적 속성관계기법은 의료영상의 해부학적 단위객체의 복잡도가 증가하여도 의료영상을 효과적으로 분류할 수 있다는 것을 여러 실험을 통해 확인하였다.
References
-
X. Li, H. Chen, X. Qi, Q. Dou, C.-W. Fu, and P.-A. Heng, “H-DenseUNet: Hybrid densely connected UNet for liver and tumor segmentation from CT volumes,” IEEE Trans. Med. Imag., Vol. 37, No. 12, pp. 2663–2674, Dec. 2018.
[https://doi.org/10.1109/TMI.2018.2845918]

-
S. Pereira, A. Pinto, V. Alves, and C. A. Silva, ‘‘Brain tumor segmentation using convolutional neural networks in MRI images,’’ IEEE Trans. Med. Imag., Vol. 35, No. 5, pp. 1240–1251, May 2016.
[https://doi.org/10.1109/TMI.2016.2538465]

-
M. Havaei, A. Davy, D. Warde-Farley, A. Biard, A. Courville, Y. Bengio, C. Pal, P.-M. Jodoin, and H. Larochelle, ‘‘Brain tumor segmentation with deep neural networks,’’ Med. Image Anal., Vol. 35, pp. 18–31, Jan. 2017.
[https://doi.org/10.1016/j.media.2016.05.004]

- B. H. Menze et al., “The multimodal brain tumor image segmentation benchmark (BRATS),” IEEE Trans. Med. Imag., Vol. 34, No. 10, pp. 1993– 2024, Oct. 2015.
-
J. Zhang, Y. Xie, Y. Xia, and C. Shen, ‘‘Attention residual learning for skin lesion classification,’’ IEEE Trans. Med. Imag., Vol. 38, No. 9, pp. 2092–2103, Sep. 2019.
[https://doi.org/10.1109/TMI.2019.2893944]

-
X. Zhao, Y. Wu, G. Song, Z. Li, Y. Zhang, and Y. Fan, ‘‘A deep learning model integrating FCNNs and CRFs for brain tumor segmentation,’’ Med. Image Anal., Vol. 43, pp. 98–111, Jan. 2018.
[https://doi.org/10.1016/j.media.2017.10.002]

-
M. Sajjad, S. Khan, K. Muhammad, W. Wu, A. Ullah, and S. W. Baik, ‘‘Multi-grade brain tumor classification using deep CNN with extensive data augmentation,’’ J. Comput. Sci., Vol. 30, pp. 174–182, Jan. 2019
[https://doi.org/10.1016/j.jocs.2018.12.003]

-
Y. Gu, X. Lu, L. Yang, B. Zhang, D. Yu, Y. Zhao, and T. Zhou, ‘‘Automatic lung nodule detection using a 3D deep convolutional neural network combined with a multi-scale prediction strategy in chest CTs,’’ Comput,’’ Comput. Biol. Med., Vol. 103, pp. 220–231, Dec. 2018.
[https://doi.org/10.1016/j.compbiomed.2018.10.011]

-
M. Yousefi, A. Krzyżak, and C. Y. Suen, ‘‘Mass detection in digi- tal breast tomosynthesis data using convolutional neural networks and multiple instance learning,’’ Comput. Biol. Med., Vol. 96, pp. 283–293, May 2018.
[https://doi.org/10.1016/j.compbiomed.2018.04.004]

-
O. Charron, A. Lallement, D. Jarnet, V. Noblet, J. B. Clavier, and P. Meyer, ‘‘Automatic detection and segmentation of brain metastases on multimodal MR images with a deep convolutional neural network,’’ Comput. Biol. Med., Vol. 95, pp. 43–54, Apr. 2018.
[https://doi.org/10.1016/j.compbiomed.2018.02.004]

-
A. Kabir Anaraki, M. Ayati, and F. Kazemi, ‘‘Magnetic resonance imaging- based brain tumor grades classification and grading via convolutional neu- ral networks and genetic algorithms,’’ Biocybern. Biomed. Eng., Vol. 39, No. 1, pp. 63–74, Jan. 2019.
[https://doi.org/10.1016/j.bbe.2018.10.004]

-
S. Deepak and P. M. Ameer, ‘‘Brain tumor classification using deep CNN features via transfer learning,’’ Comput. Biol. Med., Vol. 111, Aug. 2019.
[https://doi.org/10.1016/j.compbiomed.2019.103345]

-
L. Zhou, Z. Zhang, Y. C. Chen, Z. Y. Zhao, X. D. Yin, and H. B. Jiang, ‘‘A deep learning-based radiomics model for differentiating benign and malignant renal tumors,’’ Transl. Oncol., Vol. 12, No. 2, pp. 292–300, 2019.
[https://doi.org/10.1016/j.tranon.2018.10.012]

-
K. V. A. Muneer, V. R. Rajendran, and J. K. Paul, ‘‘Glioma tumor grade identification using artificial intelligent techniques,’’ J. Med. Syst., Vol. 43, No. 5, p. 113, Mar. 2019.
[https://doi.org/10.1007/s10916-019-1228-2]

- Q. Li, Z. Gao, and Q. Wang, ‘‘Glioma segmentation with a uni- fied algorithm in multimodal MRI images,’’ IEEE Access, Vol. 6, pp. 9543-9553, 2018.
-
V. P. Ananthi, P. Balasubramaniam, and T. Kalaiselvi, ‘‘A new fuzzy clustering algorithm for the segmentation of brain Tumor,’’ Soft Comput., Vol. 20, No. 12, pp. 4859–4879, 2016.
[https://doi.org/10.1007/s00500-015-1775-5]

저자소개

2001년 : 동국대학교 컴퓨터공학과 (공학석사)
2004년 : 동국대학교 컴퓨터공학과 (공학박사)
1996년~1998년: (주)경기은행
2005년~2006년: 동국대학교 컴퓨터공학과 IT교수
2007년~2018년: 나사렛대학교 IT융합학부 교수
2019년~현 재: 명지대학교 융합소프트웨어학부 객원교수
※관심분야:인공지능(Artificial Intelligence), 빅데이터Big Data), 의료정보(Medical Information), 기계학습(Machine Learning), 자율이동로봇(Autonomous Mobile Robot), 게임인공지능(Game AI) 등

2000년 : 동국대학교 컴퓨터교육 (교육학석사)
2009년 : 동국대학교 정보통신공학과 (공학박사)
1996년~1999년: ㈜유니파이코리아
2001년~현 재: 한국폴리텍대학 인천캠퍼스 컴퓨터정보과 교수
※관심분야:인공지능(Artificial Intelligence), 의료정보(Medical Information), 빅데이터(Big Data), 정보검색(Information Retrieval), 가상교육(Virtual Education), 게임인공지능(Game AI), 증강현실(Augmented Reality) 등